Micropèptid
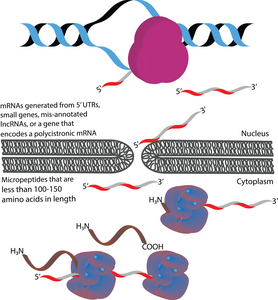
Els micropèptids (també anomenats microproteïnes) són polipèptids amb una longitud inferior a 100-150 aminoàcids que estan codificats per marcs de lectura oberts curts (sORF).[1][2][3] En aquest sentit, es diferencien de molts altres polipèptids petits actius, que es produeixen a través de la divisió postraduccional de polipèptids més grans.[1] [4] Pel que fa a la mida, els micropèptids són considerablement més curts que les proteïnes "canòniques", que tenen una longitud mitjana de 330 i 449 aminoàcids en procariotes i eucariotes, respectivament.[5] Els micropèptids de vegades s'anomenen segons la seva ubicació genòmica. Per exemple, el producte traduït d'un marc de lectura obert aigües amunt (uORF) es podria anomenar pèptid codificat per uORF (uPEP).[6] Els micropèptids no tenen seqüències de senyalització N-terminals, cosa que suggereix que és probable que estiguin localitzats al citoplasma.[1] No obstant això, alguns micropèptids s'han trobat en altres compartiments cel·lulars, tal com indica l'existència de micropèptids transmembrana.[7][8] Es troben tant en procariotes com en eucariotes.[1] [9][10] Els sORF a partir dels quals es tradueixen els micropèptids es poden codificar en UTR 5', gens petits o ARNm policistrònics. Alguns gens que codificaven micropèptids van ser originalment anotats incorrectament com a ARN llargs no codificants (lncRNAs).[11]
Donada la seva petita mida, originalment es van passar per alt els SORF. Tanmateix, s'han identificat centenars de milers de micropèptids putatius mitjançant diverses tècniques en multitud d'organismes. Només una petita fracció d'aquests amb potencial de codificació s'ha confirmat la seva expressió i funció. Els que s'han caracteritzat funcionalment, en general, tenen papers en la senyalització cel·lular, l'organogènesi i la fisiologia cel·lular. A mesura que es descobreixen més micropèptids, també ho són més les seves funcions. Una funció reguladora és la dels peptoswitches, que inhibeixen l'expressió de seqüències de codificació aigües avall mitjançant l'aturada dels ribosomes, mitjançant la seva activació directa o indirecta per molècules petites.[12]
Identificació
[modifica]Existeixen diverses tècniques experimentals per identificar els ORF potencials i els seus productes de traducció. Aquestes tècniques només són útils per a la identificació de sORF que poden produir micropèptids i no per a la caracterització funcional directa.
Seqüenciació de l'ARN
[modifica]Un mètode per trobar potencials sORF, i per tant micropèptids, és mitjançant la seqüenciació d'ARN (RNA-Seq). RNA-Seq utilitza la seqüenciació de nova generació (NGS) per determinar quins ARN s'expressen en una cèl·lula, teixit o organisme determinats en un moment concret. Aquesta col·lecció de dades, coneguda com a transcriptoma, es pot utilitzar com a recurs per trobar possibles sORF.[1] A causa de la gran probabilitat que sORF inferiors a 100 aa es produeixin per casualitat, calen estudis posteriors per determinar la validesa de les dades obtingudes mitjançant aquest mètode.[12]
Perfil de ribosomes (Ribo-Seq)
[modifica]El perfil de ribosomes s'ha utilitzat per identificar micropèptids potencials en un nombre creixent d'organismes, incloses les mosques de la fruita, el peix zebra, els ratolins i els humans.[12] Un mètode utilitza compostos com harringtonina, puromicina o lactimidomicina per aturar els ribosomes als llocs d'inici de la traducció.[13] Això indica on s'està duent a terme la traducció activa. Els inhibidors de l'allargament de la traducció, com l'emetina o la cicloheximida, també es poden utilitzar per obtenir petjades de ribosomes que tenen més probabilitats de donar lloc a un ORF traduït.[14] Si un ribosoma s'uneix a o prop d'un sORF, suposadament codifica un micropèptid.[1] [15] [16]
Espectrometria de masses
[modifica]L'espectrometria de masses (MS) és l'estàndard d'or per identificar i seqüenciar proteïnes. Mitjançant aquesta tècnica, els investigadors poden determinar si els polipèptids es tradueixen, de fet, a partir d'un sORF.
Aplicacions proteogenòmica
[modifica]La proteogenòmica combina proteòmica, genòmica i transciptòmica. Això és important quan es busquen micropèptids potencials. Un mètode per utilitzar la proteogenòmica consisteix a utilitzar dades d'ARN-Seq per crear una base de dades personalitzada de tots els polipèptids possibles. Es realitza una cromatografia líquida seguida de MS en tàndem (LC-MS/MS) per proporcionar informació de la seqüència dels productes de traducció. La comparació de les dades transcriptòmiques i proteòmiques es pot utilitzar per confirmar la presència de micropèptids.[1] [15]
Conservació filogenètica
[modifica]La conservació filogenètica pot ser una eina útil, especialment quan es revisa una gran base de dades de sORF. La probabilitat que un sORF doni lloc a un micropèptid funcional és més probable si es conserva en nombroses espècies.[12][17] Tanmateix, això no funcionarà per a tots els sORF. Per exemple, els que estan codificats per lncRNA tenen menys probabilitats de conservar-se donat que els lncRNA no tenen una alta conservació de la seqüència.[15] Seran necessaris més experimentacions per determinar si realment es produeix un micropèptid funcional.
Validació del potencial de codificació de proteïnes
[modifica]Anticossos
[modifica]Els anticossos personalitzats dirigits al micropèptid d'interès poden ser útils per quantificar l'expressió o determinar la localització intracel·lular. Com passa amb la majoria de proteïnes, la baixa expressió pot dificultar la detecció. La petita mida del micropèptid també pot comportar dificultats per dissenyar un epítop des del qual dirigir l'anticossos.[15]
Etiquetatge amb CRISPR-Cas9
[modifica]L'edició del genoma es pot utilitzar per afegir FLAG/MYC o altres etiquetes peptídiques petites a un sORF endògen, creant així proteïnes de fusió. En la majoria dels casos, aquest mètode és beneficiós perquè es pot realitzar més ràpidament que el desenvolupament d'un anticòs personalitzat. També és útil per a micropèptids per als quals no es pot dirigir cap epítop.[18]
Traducció in vitro
[modifica]Aquest procés implica clonar l'ADNc del micropèptid de longitud completa en un plasmidi que conté un promotor T7 o SP6. Aquest mètode utilitza un sistema de síntesi de proteïnes lliure de cèl·lules en presència de 35 S-metionina per produir el pèptid d'interès. A continuació, els productes es poden analitzar mitjançant electroforesi en gel i el pèptid marcat amb 35 S es visualitza mitjançant autorradiografia.[15]
Bases de dades i repositoris
[modifica]Hi ha diversos dipòsits i bases de dades que s'han creat tant per a sORF com per a micropèptids. A sORFs.org es pot trobar un repositori de petits ORF descoberts mitjançant el perfil de ribosomes.[19][20] Es pot trobar un dipòsit de pèptids putatius codificats per sORF a Arabidopsis thaliana als ARA-PEP.[21][22] A SmProt es pot trobar una base de dades de proteïnes petites, especialment codificades per ARN no codificants.[23][24]
Exemples procariotes
[modifica]Fins ara, la majoria de micropèptids s'han identificat en organismes procariotes. Tot i que la majoria encara no s'han caracteritzat completament, dels que s'han estudiat, molts semblen ser crítics per a la supervivència d'aquests organismes. A causa de la seva petita mida, els procariotes són especialment susceptibles als canvis en el seu entorn, i com a tals han desenvolupat mètodes per assegurar la seva existència.
Escherichia coli (E. coli)
[modifica]Els micropèptids expressats en E. coli exemplifiquen les adaptacions ambientals bacterianes. La majoria d'aquests s'han classificat en tres grups: pèptids líders, proteïnes ribosòmiques i proteïnes tòxiques. Les proteïnes líder regulen la transcripció i/o la traducció de proteïnes implicades en el metabolisme dels aminoàcids quan els aminoàcids són escassos. Les proteïnes ribosòmiques inclouen L36 (rpmJ) i L34 (rpmH), dos components de la subunitat ribosòmica 50S. Les proteïnes tòxiques, com ldrD, són tòxiques a nivells elevats i poden matar cèl·lules o inhibir el creixement, que funciona per reduir la viabilitat de la cèl·lula hoste.[25]
Salmonella enterica (S. enterica)
[modifica]A S. enterica, el factor de virulència MgtC està implicat en l'adaptació a entorns baixos en magnesi. El pèptid hidrofòbic MgrR, s'uneix a MgtC, provocant la seva degradació per la proteasa FtsH.[26]
Bacillus subtilis (B. subtilis)
[modifica]El micropèptid 46 aa Sda, expressat per B. subtilis, reprimeix l'esporulació quan l'inici de la replicació està deteriorat. En inhibir la histidina quinasa KinA, Sda impedeix l'activació del factor de transcripció Spo0A, que és necessari per a l'esporulació.[27]
Staphylococcus aureus (S. aureus)
[modifica]A S. aureus, hi ha un grup de micropèptids, 20-22 aa, que s'excreten durant la infecció de l'hoste per trencar les membranes de neutròfils i provocar la lisi cel·lular. Aquests micropèptids permeten al bacteri evitar la degradació per les principals defenses del sistema immunitari humà.[28][29]
Exemples eucariotes
[modifica]S'han descobert micropèptids en organismes eucariotes des de l'Arabidopsis thaliana fins als humans.
Tenen diversos papers en el desenvolupament de teixits i òrgans, així com en el manteniment i la funció un cop desenvolupats completament. Tot i que molts encara no s'han caracteritzat funcionalment, i probablement encara queden més per descobrir, a continuació es mostra un resum de les funcions dels micropèptids eucariotes identificades recentment.
Arabidopsis thaliana (A. thaliana)
[modifica]El gen POLARIS (PLS) codifica un micropèptid de 36 aa. És necessari per a un model adequat de les fulles vasculars i l'expansió cel·lular a l'arrel. Aquest micropèptid interacciona amb les proteïnes PIN del desenvolupament per formar una xarxa crítica per a la diafonia hormonal entre l'auxina, l'etilè i la citoquinina.[30][31][32]
ROTUNDIFOLIA (ROT4) a A. thaliana codifica un pèptid de 53 aa, que es localitza a la membrana plasmàtica de les cèl·lules de la fulla. El mecanisme de la funció ROT4 no s'entén bé, però els mutants tenen fulles arrodonides curtes, cosa que indica que aquest pèptid pot ser important en la morfogènesi de les fulles.[33]
Zea mays (Z. mays)
[modifica]Brick1 (Brk1) codifica un micropèptid de 76 aa, que està molt conservat tant en plantes com en animals. A Z. mays, es va trobar que estava implicat en la morfogènesi de l'epiteli foliar, en promoure múltiples esdeveniments de polarització cel·lular dependents de l'actina a l'epidermis foliar en desenvolupament.[34] Zm401p10 és un micropèptid de 89 aa, que té un paper en el desenvolupament normal del pol·len al tapetum. Després de la mitosi també és essencial en la degradació del tapetum.[35] Zm908p11 és un micropèptid de 97 aa de longitud, codificat pel gen Zm908 que s'expressa en grans de pol·len madurs. Es localitza al citoplasma dels tubs pol·línics, on ajuda al seu creixement i desenvolupament.[36]
Drosophila melanogaster (D. melanogaster)
[modifica]El gen de l'arròs polit (pri) conservat evolutivament, conegut com a tarsal-less (tal) a D. melanogaster, està implicat en la diferenciació epidèrmica. Aquesta transcripció policistrònica codifica quatre pèptids similars, que oscil·len entre 11 i 32 aa de longitud. Funcionen per truncar el factor de transcripció Shavenbaby (Svb). Això converteix Svb en un activador que regula directament l'expressió d'efectors diana, incloent miniatura (m) i shavenoid (sha), que junts són responsables de la formació del tricoma.[37]
Danio rerio (D. rerio)
[modifica]El gen Elabela (Ela) (també conegut com Apela,) és important per a l'embriogènesi.[38] S'expressa específicament durant les etapes tardanes de blàstula i gàstrula. Durant la gastrulació, és fonamental per afavorir la internalització i el moviment dirigit pel pol animal de les cèl·lules endodèrmiques mes. Després de la gastrulació, Ela s'expressa al mesoderm lateral, endoderm, així com a la notocorda anterior i posterior. Tot i que es va anotar com a lncRNA en peixos zebra, ratolí i humans, es va trobar que l'ORF 58-aa estava molt conservat entre les espècies de vertebrats. Ela es processa mitjançant l'eliminació del seu pèptid senyal N-terminal i després es secreta a l'espai extracel·lular . El seu pèptid madur 34-aa serveix com a primer lligand endogen a un GPCR conegut com a receptor d'Apelin.[39][38] La inactivació genètica d'Ela o Aplnr en el peix zebra dona lloc a fenotips sense cor.[40][41]
Mus musculus (M. musculus)
[modifica]La mioregulina (Mln) està codificada per un gen originalment anotat com a lncRNA. Mln s'expressa en els 3 tipus de múscul esquelètic i funciona de manera similar als micropèptids fosfolamban (Pln) al múscul cardíac i sarcolipina (Sln) al múscul esquelètic lent (tipus I). Aquests micropèptids interaccionen amb el reticle sarcoplasmàtic Ca <sup id="mw9Q">2+</sup> -ATPasa (SERCA), una bomba de membrana encarregada de regular l'absorció de Ca 2+ al reticle sarcoplasmàtic (SR). En inhibir la captació de Ca 2+ a la SR, provoquen relaxació muscular. De la mateixa manera, els gens d'endoregulina (ELN) i d'una altra regla (ALN) codifiquen micropèptids transmembrana que contenen el motiu d'unió SERCA i es conserven en mamífers.[42]
Myomixer (Mymx) està codificat pel gen Gm7325, un pèptid específic del múscul, de 84 aa de longitud, que té un paper durant l'embriogènesi en la fusió i la formació del múscul esquelètic. Es localitza a la membrana plasmàtica, associant-se a una proteïna de membrana fusogènica, Myomaker (Mymk). En humans, el gen que codifica Mymx s'anota com a LOC101929726 no caracteritzat. Els ortòlegs també es troben en els genomes de tortugues, granotes i peixos.[43]
Homo sapiens (H. sapiens)
[modifica]En humans, es va descobrir NoBody (polipèptid dissociador del cos P no anotat), un micropèptid de 68 aa, en l' ARN no codificant (lincRNA) LINC01420 de llarga intervenció. Té una alta conservació de seqüències entre mamífers i es localitza als cossos P. Enriqueix les proteïnes associades amb el decapsulament de l'ARNm 5' . Es creu que interactua directament amb Enhancer of mRNA Decapping 4 (EDC4).[44]
ELABELA (ELA) (també conegut com APELA) és una hormona endògena que és secretada com a micropèptid de 32 aminoàcids per les cèl·lules mare embrionàries humanes.[45] És essencial mantenir l'auto-renovació i la pluripotència de les cèl·lules mare embrionàries humanes. Els seus senyals de manera autòcrina a través de la via PI3/AKT mitjançant un receptor de superfície cel·lular encara no identificat.[46] En diferenciar les cèl·lules mesodèrmiques, l'ELA s'uneix a l'APLNR, un GPCR que també pot respondre al pèptid hormonal APLN, i fa senyals a través d'aquest.
Es preveu que el gen C7orf49, conservat en mamífers, quan s'empalma alternativament produeixi tres micropèptids. Abans es va trobar que la MRI-1 era un modulador de la infecció per retrovirus. El segon micropèptid previst, MRI-2, pot ser important en la unió d'extrems no homòlegs (NHEJ) de trencaments de doble cadena d'ADN. En els experiments de co-immunoprecipitació, la MRI-2 es va unir a Ku70 i Ku80, dues subunitats de Ku, que tenen un paper important en la via NHEJ.[47]
El micropèptid de 24 aminoàcids, Humanin (HN), interacciona amb la proteïna X associada a Bcl2 (Bax) que indueix l'apoptosi. En el seu estat actiu, Bax experimenta un canvi conformacional que exposa els dominis dirigits a la membrana. Això fa que es mogui del citosol a la membrana mitocondrial, on insereix i allibera proteïnes apoptogèniques com el citocrom c. En interactuar amb Bax, HN impedeix l'orientació de Bax als mitocondris, bloquejant així l'apoptosi.[48]
Es va trobar que un micropèptid de 90aa, ' Small Regulatory Polypeptide of Aminocid Response ' o SPAAR, estava codificat al lncRNA LINC00961 . Es conserva entre l'home i el ratolí, i es localitza a l'endosoma/lisosoma tardà. SPAAR interacciona amb quatre subunitats del complex v-ATPasa, inhibint la translocació de mTORC1 a la superfície lisosomal on s'activa. La regulació a la baixa d'aquest micropèptid permet l'activació de mTORC1 mitjançant l'estimulació d'aminoàcids, afavorint la regeneració muscular.[49]
Referències
[modifica]- ↑ 1,0 1,1 1,2 1,3 1,4 1,5 1,6 EuPA Open Proteomics, 3, 2014, pàg. 128–137. DOI: 10.1016/j.euprot.2014.02.006 [Consulta: free].
- ↑ Trends in Cell Biology, 27, 9, 9-2017, pàg. 685–696. DOI: 10.1016/j.tcb.2017.04.006. PMC: 5565689. PMID: 28528987.
- ↑ Frontiers in Plant Science, 4, 2013, pàg. 208. DOI: 10.3389/fpls.2013.00208. PMC: 3687714. PMID: 23802007 [Consulta: free].
- ↑ Development, Growth & Differentiation, 50 Suppl 1, 6-2008, pàg. S269–76. DOI: 10.1111/j.1440-169x.2008.00994.x. PMID: 18459982 [Consulta: free].
- ↑ Trends in Genetics, 16, 3, 3-2000, pàg. 107–9. DOI: 10.1016/s0168-9525(99)01922-8. PMID: 10689349.
- ↑ Proteomics, 18, 10, 5-2018, pàg. e1700035. DOI: 10.1002/pmic.201700035. PMID: 29691985 [Consulta: free].
- ↑ Cell, 160, 4, 2-2015, pàg. 595–606. DOI: 10.1016/j.cell.2015.01.009. PMC: 4356254. PMID: 25640239.
- ↑ Science, 356, 6335, 4-2017, pàg. 323–327. Bibcode: 2017Sci...356..323B. DOI: 10.1126/science.aam9361. PMC: 5502127. PMID: 28386024.
- ↑ The EMBO Journal, 27, 3, 2-2008, pàg. 546–57. DOI: 10.1038/sj.emboj.7601983. PMC: 2241655. PMID: 18200043.
- ↑ Cell, 104, 2, 1-2001, pàg. 269–79. DOI: 10.1016/s0092-8674(01)00211-2. PMID: 11207367.
- ↑ Nature Reviews. Genetics, 15, 3, 3-2014, pàg. 193–204. DOI: 10.1038/nrg3520. PMID: 24514441.
- ↑ 12,0 12,1 12,2 12,3 Nature Reviews. Genetics, 15, 3, 3-2014, pàg. 193–204. DOI: 10.1038/nrg3520. PMID: 24514441.Andrews SJ, Rothnagel JA (March 2014). "Emerging evidence for functional peptides encoded by short open reading frames". Nature Reviews. Genetics. 15 (3): 193–204. doi:10.1038/nrg3520. PMID 24514441. S2CID 22543778.
- ↑ The EMBO Journal, 33, 9, 5-2014, pàg. 981–93. DOI: 10.1002/embj.201488411. PMC: 4193932. PMID: 24705786.
- ↑ Cell Reports, 8, 5, 9-2014, pàg. 1365–79. DOI: 10.1016/j.celrep.2014.07.045. PMC: 4216110. PMID: 25159147.
- ↑ 15,0 15,1 15,2 15,3 15,4 Trends in Cell Biology, 27, 9, 9-2017, pàg. 685–696. DOI: 10.1016/j.tcb.2017.04.006. PMC: 5565689. PMID: 28528987.Makarewich CA, Olson EN (September 2017). "Mining for Micropeptides". Trends in Cell Biology. 27 (9): 685–696. doi:10.1016/j.tcb.2017.04.006. PMC 5565689. PMID 28528987.
- ↑ Annual Review of Virology, 2, 1, 11-2015, pàg. 335–49. DOI: 10.1146/annurev-virology-100114-054854. PMID: 26958919.
- ↑ The EMBO Journal, 33, 9, 5-2014, pàg. 981–93. DOI: 10.1002/embj.201488411. PMC: 4193932. PMID: 24705786.Bazzini AA, Johnstone TG, Christiano R, Mackowiak SD, Obermayer B, Fleming ES, Vejnar CE, Lee MT, Rajewsky N, Walther TC, Giraldez AJ (May 2014). "Identification of small ORFs in vertebrates using ribosome footprinting and evolutionary conservation". The EMBO Journal. 33 (9): 981–93. doi:10.1002/embj.201488411. PMC 4193932. PMID 24705786.
- ↑ Trends in Cell Biology, 27, 9, 9-2017, pàg. 685–696. DOI: 10.1016/j.tcb.2017.04.006. PMC: 5565689. PMID: 28528987.Makarewich CA, Olson EN (September 2017). "Mining for Micropeptides". Trends in Cell Biology. 27 (9): 685–696. doi:10.1016/j.tcb.2017.04.006. PMC 5565689. PMID 28528987.
- ↑ «sORFs.org: repository of small ORFs identified by ribosome profiling» (en anglès). sorfs.org. [Consulta: 14 desembre 2018].
- ↑ Nucleic Acids Research, 44, D1, 1-2016, pàg. D324–9. DOI: 10.1093/nar/gkv1175. PMC: 4702841. PMID: 26527729.
- ↑ «ARA-PEPs: A Repository of putative sORF-encoded peptides in Arabidopsis thaliana». www.biw.kuleuven.be. [Consulta: 14 desembre 2018].
- ↑ BMC Bioinformatics, 18, 1, 1-2017, pàg. 37. DOI: 10.1186/s12859-016-1458-y. PMC: 5240266. PMID: 28095775.
- ↑ «SmProt: a database of small proteins encoded by annotated coding and non-coding RNA loci». bioinfo.ibp.ac.cn. Arxivat de l'original el 2018-12-18. [Consulta: 14 desembre 2018].
- ↑ Briefings in Bioinformatics, 19, 4, 7-2018, pàg. 636–643. DOI: 10.1093/bib/bbx005. PMID: 28137767.
- ↑ Molecular Microbiology, 70, 6, 12-2008, pàg. 1487–501. DOI: 10.1111/j.1365-2958.2008.06495.x. PMC: 2614699. PMID: 19121005.
- ↑ The EMBO Journal, 27, 3, 2-2008, pàg. 546–57. DOI: 10.1038/sj.emboj.7601983. PMC: 2241655. PMID: 18200043.Alix E, Blanc-Potard AB (February 2008). "Peptide-assisted degradation of the Salmonella MgtC virulence factor". The EMBO Journal. 27 (3): 546–57. doi:10.1038/sj.emboj.7601983. PMC 2241655. PMID 18200043.
- ↑ Cell, 104, 2, 1-2001, pàg. 269–79. DOI: 10.1016/s0092-8674(01)00211-2. PMID: 11207367.Burkholder WF, Kurtser I, Grossman AD (January 2001). "Replication initiation proteins regulate a developmental checkpoint in Bacillus subtilis". Cell. 104 (2): 269–79. doi:10.1016/s0092-8674(01)00211-2. hdl:1721.1/83916. PMID 11207367. S2CID 15048130.
- ↑ Nature Medicine, 13, 12, 12-2007, pàg. 1510–4. DOI: 10.1038/nm1656. PMID: 17994102.
- ↑ Journal of Bacteriology, 192, 1, 1-2010, pàg. 46–58. DOI: 10.1128/jb.00872-09. PMC: 2798279. PMID: 19734316.
- ↑ The Plant Cell, 14, 8, 8-2002, pàg. 1705–21. DOI: 10.1105/tpc.002618. PMC: 151460. PMID: 12172017.
- ↑ The Plant Cell, 18, 11, 11-2006, pàg. 3058–72. DOI: 10.1105/tpc.106.040790. PMC: 1693943. PMID: 17138700.
- ↑ Frontiers in Plant Science, 4, 2013, pàg. 75. DOI: 10.3389/fpls.2013.00075. PMC: 3617403. PMID: 23577016 [Consulta: free].
- ↑ The Plant Journal, 38, 4, 5-2004, pàg. 699–713. DOI: 10.1111/j.1365-313x.2004.02078.x. PMID: 15125775.
- ↑ Current Biology, 12, 10, 5-2002, pàg. 849–53. DOI: 10.1016/s0960-9822(02)00819-9. PMID: 12015123 [Consulta: free].
- ↑ Functional Plant Biology, 36, 1, 2009, pàg. 73–85. DOI: 10.1071/fp08154. PMID: 32688629.
- ↑ Journal of Experimental Botany, 64, 8, 5-2013, pàg. 2359–72. DOI: 10.1093/jxb/ert093. PMC: 3654424. PMID: 23676884.
- ↑ Science, 329, 5989, 7-2010, pàg. 336–9. Bibcode: 2010Sci...329..336K. DOI: 10.1126/science.1188158. PMID: 20647469.
- ↑ 38,0 38,1 Developmental Cell, 27, 6, 12-2013, pàg. 672–80. DOI: 10.1016/j.devcel.2013.11.002. PMID: 24316148 [Consulta: free].
- ↑ Science, 343, 6172, 2-2014, pàg. 1248636. DOI: 10.1126/science.1248636. PMC: 4107353. PMID: 24407481.
- ↑ Deshwar, Ashish R; Chng, Serene C; Ho, Lena; Reversade, Bruno; Scott, Ian C eLife, 5, 14-04-2016, pàg. e13758. DOI: 10.7554/eLife.13758. ISSN: 2050-084X. PMC: 4859801. PMID: 27077952.
- ↑ Scott, Ian C.; Masri, Bernard; D'Amico, Leonard A.; Jin, Suk-Won; Jungblut, Benno Developmental Cell, 12, 3, 3-2007, pàg. 403–413. DOI: 10.1016/j.devcel.2007.01.012. ISSN: 1534-5807. PMID: 17336906 [Consulta: free].
- ↑ Cell, 160, 4, 2-2015, pàg. 595–606. DOI: 10.1016/j.cell.2015.01.009. PMC: 4356254. PMID: 25640239.Anderson DM, Anderson KM, Chang CL, Makarewich CA, Nelson BR, McAnally JR, Kasaragod P, Shelton JM, Liou J, Bassel-Duby R, Olson EN (February 2015). "A micropeptide encoded by a putative long noncoding RNA regulates muscle performance". Cell. 160 (4): 595–606. doi:10.1016/j.cell.2015.01.009. PMC 4356254. PMID 25640239.
- ↑ Science, 356, 6335, 4-2017, pàg. 323–327. Bibcode: 2017Sci...356..323B. DOI: 10.1126/science.aam9361. PMC: 5502127. PMID: 28386024.Bi P, Ramirez-Martinez A, Li H, Cannavino J, McAnally JR, Shelton JM, Sánchez-Ortiz E, Bassel-Duby R, Olson EN (April 2017). "Control of muscle formation by the fusogenic micropeptide myomixer". Science. 356 (6335): 323–327. Bibcode:2017Sci...356..323B. doi:10.1126/science.aam9361. PMC 5502127. PMID 28386024.
- ↑ Nature Chemical Biology, 13, 2, 2-2017, pàg. 174–180. DOI: 10.1038/nchembio.2249. PMC: 5247292. PMID: 27918561.
- ↑ Developmental Cell, 27, 6, 12-2013, pàg. 672–80. DOI: 10.1016/j.devcel.2013.11.002. PMID: 24316148 [Consulta: free].Chng SC, Ho L, Tian J, Reversade B (December 2013). "ELABELA: a hormone essential for heart development signals via the apelin receptor". Developmental Cell. 27 (6): 672–80. doi:10.1016/j.devcel.2013.11.002. PMID 24316148.
- ↑ Ho, Lena; Tan, Shawn Y. X.; Wee, Sheena; Wu, Yixuan; Tan, Sam J. C. Cell Stem Cell, 17, 4, 01-10-2015, pàg. 435–447. DOI: 10.1016/j.stem.2015.08.010. ISSN: 1875-9777. PMID: 26387754 [Consulta: free].
- ↑ The Journal of Biological Chemistry, 289, 16, 4-2014, pàg. 10950–7. DOI: 10.1074/jbc.c113.533968. PMC: 4036235. PMID: 24610814 [Consulta: free].
- ↑ Nature, 423, 6938, 5-2003, pàg. 456–61. Bibcode: 2003Natur.423..456G. DOI: 10.1038/nature01627. PMID: 12732850.
- ↑ Nature, 541, 7636, 1-2017, pàg. 228–232. Bibcode: 2017Natur.541..228M. DOI: 10.1038/nature21034. PMID: 28024296.
