Triptòfan sintetasa
|
|
Aquest article o secció necessita millorar una traducció deficient. |
 Subunitats: Subunitat β, Subunitat α amb PLP, IGP | |||||||||
| Identificadors | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Número EC | 4.2.1.20 | ||||||||
| Número CAS | 9014-52-2
| ||||||||
| Bases de dades | |||||||||
| IntEnz | IntEnz view | ||||||||
| BRENDA | BRENDA entry | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | KEGG entry | ||||||||
| MetaCyc | metabolic pathway | ||||||||
| PRIAM | profile | ||||||||
| Estructures PDB | RCSB PDB PDBj PDBe PDBsum | ||||||||
| |||||||||
La triptòfan sinteteasa és un enzim que catalitza els dos passos finals en la biosíntesi del triptòfan.[1] És generalment present en eubacteris, arqueus, protistes, fongs, i plantes.[2][3][4][5][6] Però, és absent en els animals.[7] És típicament present com un tetràmer α2β2. Les subunitats α catalitzen la formació reversible de indole i gliceraldehid 3-fosfat (G3P) a partir d'indole-3-glicerolfosfat (IGP). Les subunitats β catalitzen la condensació irreversible d'indole i serina per formar triptòfan mitjançant una reacció depenent de piridoxal fosfat (PLP). Cada centre actiu d'una subunitat α ésta connectat al centre actiu d'una subunitat β per un canal hidròfob de 25 àngstroms de llarg a l'interior de l'enzim. Això facilita la difusió de l'indole format als centres actius α directament cap als centres actius β en un procés anomenat canalització de substrats.[8] Els centres actius de la triptòfan sintetasa estan acoblats al·lostèricament.[9]
Estructura
[modifica]Subunitats: La triptòfan sintetasa existeix normalment com un complex α-ββ-α. Les subunitats α i β tenen masses moleculars de 27 i 43 kDa respectivament. La subunitat α té un conformació de barril TIM. La subunitat β té un lloc d'unió per a cations monovalents que és adjacent al centre actiu.[10] L'assemblatge de les subunitats per formar el complex comporta canvis estructurals que alhora activen ambdues subunitats. Hi ha dos mecanismes principals de comunicació entre les subunitats. El domini COMM de la subunitat β i el loop 2 de la subunitat α. A més, hi ha interaccions entre els residus αGly181 i βSer178.[11] Els centres actius són regulats al·lostèricament i experimenten transicions entre els estats obert-inactiu, i tancat-actiu.[9]
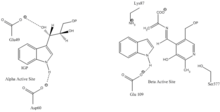
Lloc d'unió del indol-3-glicerol : Vegeu imatge 1.
Lloc d'unió d'indole i serina: Vegeu imatge 1.
Canal hidrofòbic: Els centres actius α i β estan separats per un canal hidrofòbic de 25 àngstroms de llarg a l'interior de l'enzim que permet la difusió d'indole. Si el canal no existís, l'indole format a un centre actiu α difondria ràpidament, allunyant-se de l'enzim, i es perdria en l'interior de la cèl·lula degut al fet que és hidrofòbic i pot creuar membranes fàcilment. Per això, el canal és essencial per funció de l'enzim.[12]
Mecanisme d'acció
[modifica]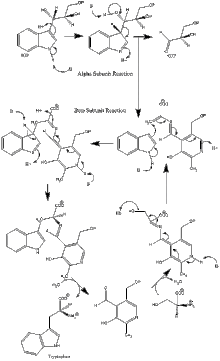
Reacció en la subunitat α: La subunitat α catalitza la formació d'indole i G3P a partir d'IGP mitjançant una reacció retro-aldòlica. Es creu que els residus αGlu49 i αAsp60 estan directament implicats en la catàlisi.[8] L'etapa limitant és la isomerització d'IGP.[13] Vegeu la imatge 2.
Reacció en la subunitat β: La subunitat β catalitza la reacció de substitució en la qual l'indole i la serina condensen per formar triptòfan en una reacció depenent de piridoxal fosfat (PLP). Es creu que els residus βLys87, βGlu109, i βSer377 estan directament implicats en la catàlisi.[8] Tanmateix, el mecanisme exacte no ha estat determinat. Vegeu imatge 2.
Reacció: Vegeu imatge 3.

Funció biològica
[modifica]El triptòfan és un dels vint-i-dos aminoàcids estàndards i un dels nou aminoàcids essencials pels éssers humans. Per això, el triptòfan és un component necessari en la dieta humana.
Rellevància clínica
[modifica]Com que els éssers humans no tenen triptòfan sintetasa, aquest enzim ha estat explorat com un possible fàrmac contra infeccions bacterianes.[14] Tanmateix, es creu que els bacteris tenen mecanismes alternatius per produir aminoàcids que podrien fer aquesta tàctica menys eficaç. En qualsevol cas, fins i tot si el fàrmac només debilita els bacteris, encara podria ser útil, ja que els bacteris són vulnerables en l'entorn hostil de l'amfitrió. Per això, la inhibició de triptòfan sintetasa juntament amb la d'altres enzims PLP involucrats en el metabolisme d'aminoàcids té el potencial d'ajudar a solucionar problemes mèdics.[15]
- Tractament de tuberculosi[14]
- Tractament d'infeccions oculars i genitals[16]
- Tractament de crjptosporidiosi[14]
- Ús herbicida[17]
Evolució
[modifica]Es creu que a l'inici de la seva evolució el gen trpB2 va ser duplicat. Una còpia, anomenada trpB2i, va passar a formar part de l'operó trp permetent la seva expressió conjunta amb el gen trpA. El producte TrpB2i formava complexos transitoris amb el TrpA activant aquest últim de manera unidireccional. L'altra còpia, anomenada trpB2o, va quedar aïllada va complint una funció prèviament existent o bé obtenint-ne una de nova com podria la retenció d'indole per part d'aquesta proteïna. El trpB2i va evolucionar a TrpB1, el qual formava complexos permanents amb trpA resultant en una activació bidireccional. L'avantatge de la retenció d'indole per part de la proteïna va declinar i el gen trpB2o es va perdre. Finalment, els gens TrpB1 i TrpA es van fusionar resultant en la formació de l'enzim bifuncional.[18]
Importància històrica
[modifica]La triptòfan sintetasa va ser el primer enzim identificat amb dues capacitats catalítiques que eren àmpliament estudiades. També va ser el primer enzim identificat que feia servir canalització de substrat. Per això, aquest enzim ha estat estudiat extensament i és un tema de gran interès.[8]
Referències
[modifica]- ↑ Dunn MF, Niks D, Ngo H, Barends TRM, Schlichting I «Tryptophan synthase: the workings of a channeling nanomachine». Trends in Biochemical Sciences, 33, 6, 6-2008, pàg. 254–64. DOI: 10.1016/j.tibs.2008.04.008. PMID: 18486479.
- ↑ Jablonski P, Jablonski L, Pintado O, Sriranganathan N, Howde C «Tryptophan synthase: Identification of Pasteurella multocida tryptophan synthase B-subunit by antisera against strain PI059». Microbiology, 142, 9-1996, pàg. 115–21. DOI: 10.1099/13500872-142-1-115. PMID: 8581158.
- ↑ Lazcano A, Diaz-Villgomez E, Mills T, Oro J «On the levels of enzymatic substrate specificity: Implications for the early evolution of metabolic pathways». Advances in Space Research, 15, 3, 3-1995, pàg. 345–56. DOI: 10.1016/S0273-1177(99)80106-9. PMID: 11539248.
- ↑ Anderson I, Watkins R, Samuelson J, Spencer D, Majoros W, Grey M, Loftus B «Gene Discovery in the Acanthamoeba castellanii Genome». Protist, 156, 2, 8-2005, pàg. 203-14. DOI: 10.1016/j.protis.2005.04.001. PMID: 16171187.
- ↑ ,Ireland C, Peekhaus N, Lu P, Sangari R, Zhang A, Masurekar P, An Z «The tryptophan synthetase gene TRP1 of Nodulisporium sp.: molecular characterization and its relation to nodulisporic acid A production». Appl Microbiol Biotechnol, 79, 3, 4-2008, pàg. 451–9. DOI: 10.1007/s00253-008-1440-3. PMID: 18389234.
- ↑ Sanjaya, Hsiao PY, Su RC, Ko SS, Tong CG, Yang RY, Chan MT «Overexpression of Arabidopsis thaliana tryptophan synthase beta 1 (AtTSB1) in Arabidopsis and tomato confers tolerance to cadmium stress». Plant Cell Environ, 31, 8, 4-2008, pàg. 1074–85. DOI: 10.1111/j.1365-3040.2008.01819.x. PMID: 18419734.
- ↑ Eckert SC, Kubler E, Hoffmann B, Braus GH «The tryptophan synthase-encoding trpB gene of Aspergillus nidulans is regulated by the cross-pathway control system». Mol Gen Genet, 263, 5, 6-2000, pàg. 867–76. DOI: 10.1007/s004380000250. PMID: 10905354.
- ↑ 8,0 8,1 8,2 8,3 Raboni S, Bettati S, Mozzarelli A «Tryptophan synthase: a mine for enzymologists». Cell Mol Life Sci, 66, 14, 4-2009, pàg. 2391–403. DOI: 10.1007/s00018-009-0028-0. PMID: 19387555.
- ↑ 9,0 9,1 Fatmi MQ, Ai R, Chang CA «Synergistic regulation and ligand-induced conformational changes of tryptophan synthase». Biochemistry, 48, 41, 9-2009, pàg. 9921–31. DOI: 10.1021/bi901358j. PMID: 19764814.
- ↑ Grishin NV, Phillips MA, Goldsmith EJ «Modeling of the spatial structure of ornithine decarboxylases». Protein Sci, 4, 7, 7-1995, pàg. 1291–304. DOI: 10.1002/pro.5560040705. PMC: 2143167. PMID: 7670372.
- ↑ Schneider TR, Gerhardt E, Lee M, Liang PH, Anderson KS, Schlichting I «Loop closure and intersubunit communication in tryptophan synthase». Biochemistry, 37, 16, 4-1998, pàg. 5394–406. DOI: 10.1021/bi9728957. PMID: 9548921.
- ↑ Huang X, Holden HM, Raushel FM «Channeling of Substrates and Intermediates in Enzyme-Catalyzes Reactions». Annu Rev Biochem, 70, 2001, pàg. 149–80. DOI: 10.1146/annurev.biochem.70.1.149. PMID: 11395405.
- ↑ Anderson KS, Miles EW, Johnson KA «Serine modulates substrate channeling in tryptophan synthase. A novel intersubunit triggering mechanism». J Biol Chem, 266, 13, 5-1991, pàg. 8020–33. PMID: 1902468.
- ↑ 14,0 14,1 14,2 Chaudhary K, Roos DS «Protozoan genomics for drug discovery». Nat Biotechnol, 23, 9, 9-2005, pàg. 1089–91. DOI: 10.1038/nbt0905-1089. PMID: 16151400.
- ↑ Becker D, Selbach M, Rollenhagen C, Ballmaier M, Meyer TF, Mann M, Bumann D «Robust Salmonella metabolism limits possibilities for new antimicrobials». Nature, 440, 7082, 3-2006, pàg. 303–7. DOI: 10.1038/nature04616. PMID: 16541065.
- ↑ Caldwell HD, Wood H, Crance D, Baily R «Polymorphisms in Chlamydia trachomatis tryptophan synthase genes differentiate between genital an ocular isolates». J Clin Invest, 111, 11, 6-2003, pàg. 1757–69. DOI: 10.1172/JCI17993. PMC: 156111. PMID: 12782678.
- ↑ Kulik V, Hartmann E, Weyand M, Frey M, Gierl A, Niks D, Dunn MF, Schlichting I «On the structural basis of the catalytic mechanism and the regulation of the α-subunit of tryptophan synthase from Salmonella typhimurium and BXI from maize, two evolutionarily related enzymes». J Mol Biol, 352, 3, 9-2005, pàg. 608–20. DOI: 10.1016/j.jmb.2005.07.014. PMID: 16120446.
- ↑ Leopoldseder S, Hettwer S, Sterner R «Evolution of Multi-Enzyme Complexes: The Case of Tryptophan Synthase». Biochemistry, 45, 47, 11-2006, pàg. 14111–9. DOI: 10.1021/bi061684b. PMID: 17115706.
