Àcid desoxiribonucleic
| «ADN» redirigeix aquí. Vegeu-ne altres significats a «ADN (desambiguació)». |
| Part d'una sèrie sobre |
| Genètica |
|---|
 |
|
Conceptes fonamentals |
|
Medicina de precisió |
L'àcid desoxiribonucleic (ADN, conegut igualment per la sigla anglesa DNA) és una molècula composta de dues cadenes de polinucleòtids enrotllades al llarg d'un eix comú que formen una doble hèlix amb les instruccions genètiques per al desenvolupament, el funcionament, el creixement i la reproducció de tots els organismes coneguts i molts virus. L'ADN i l'àcid ribonucleic (ARN) són àcids nucleics, que constitueixen els quatre tipus de macromolècules essencials per a la vida juntament amb els lípids, les proteïnes i els glúcids.
Les cadenes d'ADN són polinucleòtids, és a dir, es componen de monòmers més petits que es coneixen com a «nucleòtids».
Sovint es compara l'ADN a un conjunt de plantilles, una recepta, o un codi, car conté les instruccions requerides per produir altres components de les cèl·lules, com ara proteïnes i molècules d'àcid ribonucleic (ARN). Els segments d'ADN que porten aquesta informació genètica reben el nom de «gen», però altres seqüències d'ADN tenen una funció estructural o ajuden a regular l'ús d'aquesta informació genètica.
Des d'un punt de vista químic, l'ADN es compon de dos llargs polímers d'unitats simples anomenades nucleòtids, amb un esquelet compost de sucres i grups fosfats units per enllaços èster. Aquestes dues cadenes s'estenen en direccions oposades l'una de l'altra, i per tant són antiparal·leles. Cada sucre està unit a una d'entre quatre tipus de molècules anomenades bases: adenina (A), timina (T), citosina (C) i guanina (G). La seqüència d'aquestes quatre bases al llarg de la cadena en codifica les dades. La informació és interpretada utilitzant el codi genètic, que especifica la seqüència dels aminoàcids en les proteïnes. El codi és llegit per mitjà de la còpia de segments d'ADN en l'ARN, un àcid nucleic relacionat, en un procés anomenat «transcripció».
Dins de les cèl·lules, l'ADN s'organitza en estructures anomenades cromosomes. Aquests cromosomes són duplicats abans que les cèl·lules es divideixin, en un procés anomenat replicació de l'ADN. Els organismes eucariotes (animals, plantes, fongs i protists) emmagatzemen l'ADN dins del nucli cel·lular, mentre que en els procariotes (eubacteris i arqueus), es troba dins del citoplasma de la cèl·lula. Dins dels cromosomes, proteïnes cromatines com ara les histones compacten i organitzen l'ADN. Aquestes estructures compactes guien la interacció entre l'ADN i altres proteïnes, ajudant a controlar quines parts de l'ADN són transcrites.
Propietats químiques

L'ADN és un llarg polímer que es compon d'unitats repetides anomenades «nucleòtids».[1][2] Té una estructura no-estàtica[3] en les quals totes les espècies es componen de dues cadenes helicoïdals que es caragolen al voltant d'un mateix eix, amb un pas de 34 Å (3,4 nm) i un radi de 10 Å (1,0 nm).[4] En un altre estudi, una cadena d'ADN mesurada en una solució determinada tenia una amplada de 22–26 Å (2,2–2,6 nm), mentre que un nucleòtid tenia una llargada de 3,3 Å (0,33 nm).[5] Tot i que cada unitat repetida és molt petita, els polímers d'ADN poden ser molècules enormes que contenen milions de nucleòtids. Per exemple, el cromosoma humà més gros (cromosoma 1) es compon d'aproximadament 220 milions de parells de bases[6] i tindria una llargada de 85 mm si se l'estirés.
En els organismes vius, l'ADN no sol existir en forma de molècula única, sinó com un parell de molècules estretament associades.[7][8] Aquestes dues cadenes s'entrellacen, formant una doble hèlix. Els nucleòtids repetits contenen tant el segment de l'esquelet de la molècula, que manté la cadena unida, i una base, que interacciona amb l'altra cadena d'ADN de l'hèlix. Una base unida a un sucre s'anomena nucleòsid, i una base unida a un sucre i un o més grups fosfat rep el nom de nucleòtid. Si diversos nucleòtids estan units, com en el cas de l'ADN, el polímer rep el nom de polinucleòtid.[9]
L'esquelet de la cadena d'ADN es compon de residus de fosfats i de pentoses - sucres de cinc àtoms de carboni - que es van alternant.[10] El sucre de l'ADN és la 2-desoxiribosa. Una de les diferències principals entre l'ADN i l'ARN és el sucre; en l'ARN, la 2-desoxiribosa és substituïda pel sucre pentosa alternatiu ribosa.[8] Les pentoses s'uneixen per grups fosfat que formen enllaços fosfodièster entre el tercer i el cinquè àtom de carboni dels anells de sucres adjacents.
La doble hèlix de l'ADN és estabilitzada per enllaços dèbils, especialment enllaços d'hidrogen entre les bases unides a les dues cadenes. Les quatre bases de l'ADN són l'adenina (abreujada com a A), la citosina (C), la guanina (G) i la timina (T). Aquestes quatre bases s'uneixen al sucre/fosfat per formar el nucleòtid complet, com es mostra en el cas del monofosfat d'adenosina.
Les bases es classifiquen en dos tipus; l'adenina i la guanina són compostos heterocíclics de cinc i sis membres anomenats purines, mentre que la citosina i la timina són anells de sis membres anomenats pirimidines.[8] Una cinquena base pirimidínica, anomenada uracil (U), pren sovint el lloc de la timina en l'ARN, i se'n diferencia pel fet que manca de grup metil a l'anell. L'uracil no se sol trobar a l'ADN, existint únicament com a producte de la desfragmentació de la citosina. Per tal de mantenir el diàmetre de la doble hèlix estable les pirimidines s'uneixen a les purines i específicament G s'uneix a C i A s'uneix a T. Aquest aparellament de bases fa que les dues cadenes s'oposin una a l'altra de la mateixa manera que ho fa una cremallera.
La seqüència de nucleòtids de la cadena acostuma a anomenar-se estructura primària. Tanmateix en ser una cadena tan llarga la disposició que prenen les cadenes origina una estructura secundària que és la que es produeix per a l'empaquetament de l'ADN a l'interior de la cèl·lula.
Modificacions químiques

|

|

|
| citosina | 5-metilcitosina | timina |
Modificacions de bases
L'expressió dels gens està influenciada per la manera en què l'ADN està empaquetat als cromosomes, en una estructura anomenada cromatina. Les modificacions de bases poden estar implicades en l'empaquetament; les regions que tenen poca o cap expressió gènica solen contenir nivells elevats de metilació de bases de citosina. Per exemple, la metilació de la citosina produeix 5-metilcitosina, que és important en la inactivació del cromosoma X.[11] El nivell mitjà de metilació varia entre organismes - el cuc Caenorhabditis elegans no presenta metilació de citosina, mentre que els vertebrats tenen nivells més elevats, i fins a un 1% del seu ADN conté 5-metilcitosina.[12] Malgrat la importància de la 5-metilcitosina, es pot desaminar per formar una base de timina, de manera que les citosines metilades són especialment propenses a mutar.[13] Altres modificacions de bases inclouen la metilació de l'adenina en els eubacteris, la presència de 5-hidroximetilcitosina al cervell,[14] i la glicosilació de l'uracil per produir la «base-J» en els cinetoplàstids.[15][16]
Danys

L'ADN pot resultar danyat per molts tipus diferents de mutàgens, que canvien la seqüència de l'ADN. Els mutàgens inclouen agents oxidants, agents alquilants i radiació electromagnètica d'alta energia com ara la llum ultraviolada i els rajos X. El tipus de danys a l'ADN depèn del tipus de mutagen. Per exemple, la llum ultraviolada pot danyar l'ADN produint dímers de timina, que són reticulacions entre bases de pirimidina.[18] D'altra banda, oxidants com ara els radicals lliures o el peròxid d'hidrogen causen múltiples formes de danys, incloent-hi modificacions de bases (particularment de guanosina) i trencaments de la cadena doble.[19] Una cèl·lula humana típica conté unes 150.000 bases que han patit danys oxidatius.[20] D'aquestes lesions oxidatives, les més perilloses són els trencaments de la cadena doble, car resulten difícils de reparar i poden produir mutuacions puntuals, insercions i delecions de la seqüència d'ADN, així com translocacions cromosòmiques.[21]
Molts mutàgens s'insereixen a l'espai entre dos parells de bases adjacents, un fenomen anomenat intercalació. La majoria d'intercaladors són molècules planars i aromàtiques, i inclouen el bromur d'etidi, la daunomicina i la doxorubicina. Perquè un intercalador es pugui inserir entre els parells de bases, cal que se separin les bases, distorsionant les cadenes d'ADN pel desenrotllament de la doble hèlix. Això inhibeix tant la transcripció com la replicació de l'ADN, provocant toxicitat i mutacions. Com a resultat, els intercaladors d'ADN sovint són carcinògens; l'epòxid diol de benzo[a]pirè, les acridines, les aflatoxines i el bromur d'etidi en són exemples coneguts.[22][23][24] Tanmateix, a causa de la seva capacitat d'inhibir la transcripció i la replicació de l'ADN, altres toxines similars també es fan servir en la quimioteràpia per inhibir les cèl·lules canceroses en creixement ràpid.[25]
Funcions biològiques
L'ADN existeix habitualment en forma de cromosomes linears en els eucariotes, i circulars en els procariotes. El conjunt de cromosomes d'una cèl·lula forma el seu genoma; el genoma humà té aproximadament 3.000 milions de parells de bases d'ADN arranjats en 46 cromosomes.[26] La informació emmagatzemada a l'ADN es troba a les seqüències de peces d'ADN anomenades gens. La transmissió de la informació genètica dels gens es fa per aparellemant de bases complementàries. Per exemple, en la transcripció, quan una cèl·lula utilitza la informació d'un gen, la seqüència d'ADN és copiada a una seqüència complementària d'ARN per mitjà de l'atracció entre l'ADN i els nucleòtids d'ARN adequats. Habitualment, aquesta còpia d'ARN és utilitzada per generar una seqüència proteica corresponent en un procés anomenat traducció que depèn de la mateixa interacció entre nucleòtids d'ARN. Alternativament, una cèl·lula pot simplement copiar la seva informació genètica en un procés anomenat replicació de l'ADN. Els detalls d'aquestes funcions estan explicats en altres articles; aquest es concentra en les interaccions entre l'ADN i les altres molècules que medien el funcionament del genoma.
Gens i genomes
L'ADN genòmic és empaquetat de manera compacta i endreçada per un procés conegut com a «condensació de l'ADN», que l'adapta al petit volum de l'espai intracel·lular. Els eucariotes conserven el seu ADN al nucli cel·lular, i, en quantitats baixes, als mitocondris i cloroplasts. En els procariotes, l'ADN es troba al nucleoide, un cos de forma irregular situat al citoplasma.[27] Els gens contenen la informació genètica encabida en un genoma, mentre que el conjunt complet de dades dins un organisme és el seu genotip. Un gen és una unitat d'herència i és una regió de l'ADN que influeix un caràcter determinat en un organisme. Els gens contenen una pauta oberta de lectura que es pot transcriure, així com seqüències reguladores com ara promotors i estimuladors, que controlen la transcripció de la pauta oberta de lectura.
En moltes espècies, només una fracció petita de tot el genoma codifica proteïnes. Per exemple, només un 1,5% del genoma humà es compon d'exons codificadors de proteïnes, i més del 50% de l'ADN humà es compon de seqüències repetides no codificants.[28] Les raons darrere la presència de tant ADN no codificant als genomes eucariotes i les extraordinàries diferències en la mida del genoma, o valor C, entre espècies representen un trencaclosques que ve de fa molt de temps, conegut com a paradoxa del valor C.[29] Tanmateix, les seqüències d'ADN que no codifiquen proteïnes encara poden codificar molècules funcionals d'ARN no codificant, que participen en la regulació de l'expressió gènica.[30]

Algunes seqüències d'ADN no codificant tenen un paper estructural als cromosomes. Els telòmers i centròmers típicament contenen pocs gens, però són importants pel funcionament i l'estabilitat dels cromosomes.[32][33] Una forma abundant d'ADN no codificant en els humans són els pseudogens, que són còpies de gens que han estat desactivats per mutació.[34] Sovint aquestes seqüències no són més que fòssils moleculars, tot i que a vegades serveixen com a matèria primera genètica per crear nous gens a través del procés de duplicació i divergència gènica.[35]
Transcripció i traducció
Un gen és una seqüència d'ADN que conté informació genètica i pot influir en el fenotip d'un organisme. Dins d'un gen, la seqüència de bases al llarg d'una cadena d'ADN defineix una seqüència d'ARN missatger, que després defineix una o més seqüències de proteïnes. La relació entre les seqüències de nucleòtids dels gens i les seqüències d'aminoàcids de les proteïnes ve determinada per les regles de la traducció, conegudes col·lectivament com a codi genètic. El codi genètic es compon de "paraules" de tres lletres anomenades codons, formades a partir d'una seqüència de tres nuclèotids (p. ex.: ACT, CAG, TTT).
En la transcripció, els codons d'un gen són copiats a l'ARN missatge per l'ARN polimerasa. Aquesta còpia d'ARN és posteriorment descodificada per un ribosoma que llegeix la seqüència d'ARN aparellant les bases de l'ARN missatger a l'ARN de transferència, que porta aminoàcids. Com que hi ha quatre bases en combinacions de tres lletres, hi ha 64 codons possibles ( combinacions). Aquests codons codifiquen els vint aminoàcids estàndard, donant més d'un codó possible a la majoria d'aminoàcids. També hi ha tres codons "stop" o "sense sentit" que signifiquen que s'ha arribat a la fi de la regió codificant; són els codons TAA, TGA i TAG.
El codi genètic és universal: tot i que hi ha algunes excepcions (principalment entre els protozous) en totes les espècies cada codó es tradueix pel mateix aminoàcid. Això té especial importància, ja que és el que es permet que es puguin fer transgènics (on gens d'una espècie s'insereixen en una altra).

Replicació
La divisió cel·lular és essencial pel creixement d'un organisme, però quan una cèl·lula es divideix li cal replicar l'ADN del seu genoma per tal que les dues cèl·lules filles tinguin la mateixa informació genètica que la mare. L'estructura bicatenària de l'ADN té un mecanisme simple per la replicació de l'ADN. Les dues cadenes són separades i aleshores un enzim anomenat ADN polimerasa recrea la seqüència d'ADN complementari de cada cadena. Aquest enzim crea la cadena complementària trobant la base correcta per mitjà d'aparellament de bases complementàries, i unint-la a la cadena original. Com que les ADN polimerases només poden allargar una cadena d'ADN en una direcció de 5′ a 3′, s'utilitzen diferents mecanismes per copiar les cadenes antiparal·leles de la doble hèlix.[36] D'aquesta manera, la base de la cadena inicial determina la base que apareixerà a la cadena nova, i la cèl·lula acaba amb una còpia perfecta del seu ADN.
Interacció amb les proteïnes
Totes les funcions de l'ADN depenen de les interaccions amb les proteïnes. Aquestes interaccions proteiques poden ser no específiques, o la proteïna es pot unir específicament a una sola seqüència d'ADN. Els enzims també es poden unir a l'ADN i, entre ells, les polimerases que copien la seqüència de bases d'ADN en la transcripció i la replicació de l'ADN són especialment importants.
Proteïnes d'unió a ADN
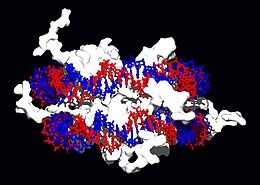
|
Les proteïnes estructurals que s'uneixen a l'ADN són exemples ben compresos d'interaccions no específiques entre ADN i proteïnes. Dins dels cromosomes, l'ADN es troba dins de complexos amb proteïnes estructurals. Aquestes proteïnes organitzen l'ADN en una estructura compacta anomenada cromatina. En els eucariotes, aquesta estructura implica la unió de l'ADN amb un complex de petites proteïnes bàsiques anomenades histones, mentre que en els procariotes hi estan implicats múltiples tipus de proteïnes.[37][38] Les histones formen un complex en forma de disc anomenat nucleosoma, que conté dues capes completes d'ADN bicatenari embolcallant-ne la superfície. Aquestes interaccions no específiques es formen quan residus bàsics de les histones formen enllaços iònics amb l'esquelet sucre-fosfat àcid de l'ADN, i per tant són en gran manera independents de la seqüència de bases.[39] Les modificacions químiques d'aquests residus d'aminoàcids bàsics inclouen la metilació, la fosforilació i l'acetilació.[40] Aquests canvis químics alteren la força de la interacció entre l'ADN i les histones, fent que l'ADN sigui més o menys accessible pels factors de transcripció i canviant la velocitat de transcripció.[41] Altres proteïnes d'unió a ADN no específiques de la cromatina inclouen les proteïnes del grup d'alta mobilitat, que s'uneixen a ADN tort o distorsionat.[42] Aquestes proteïnes són importants en la flexió de les fileres de nucleosomes i els endrecen en estructures més grans que fan els cromosomes.[43]
Un grup distint de proteïnes d'unió a ADN són les que s'uneixen específicament a l'ADN monocatenari. En els humans, la proteïna de la replicació A és el membre més ben comprès d'aquesta família i se la fa servir en processos en què se separa la doble hèlix, incloent-hi la replicació, la reparació i la recombinació de l'ADN.[44] Sembla que aquestes proteïnes d'unió estabilitzen l'ADN monocatenari i eviten que formi llaços o sigui degradat per nucleases.

En canvi, altres proteïnes han evolucionat per unir-se a determinades seqüències d'ADN. Entre elles, les que han estat estudiades més intensament són els diversos factors de transcripció, que són proteïnes que regulen la transcripció. Cada factor de transcripció s'uneix a un conjunt particular de seqüències d'ADN i activa o inhibeix la transcripció dels gens que tenen aquestes seqüències a prop dels promotors. Els factors de transcripció fan això de dues maneres. Primerament, es poden unir a l'ARN polimerasa encarregada de la transcripció, o bé directament o bé a través d'altres proteïnes mediadores; això situa la polimerasa al promotor i li permet iniciar la transcripció.[46] Alternativament, els factors de transcripció poden unir-se a enzims que modifiquen les histones del promotor; això canvia l'accessibilitat de la plantilla de l'ADN per la polimerasa.[47]
Com que aquestes dianes d'ADN poden trobar-se arreu del genoma d'un organisme, els canvis en l'activitat d'un tipus de factor de transcripció pot afectar milers de gens.[48] Per consegüent, aquestes proteïnes són sovint la diana dels processos de transducció de senyals que controlen les respostes als canvis ambientals o la diferenciació i el desenvolupament cel·lulars. L'especificitat de les interaccions d'aquests factors de transcripció amb l'ADN deriva del fet que les proteïnes fan múltiples contactes amb les vores de les bases de l'ADN, permetent-los "llegir" la seqüència d'ADN. La majoria d'aquestes interaccions amb les bases es fan al solc major, on les bases són més accessibles.[49]

Enzims modificadors de l'ADN
Nucleases i ligases
Les nucleases són enzims que tallen cadenes d'ADN catalitzant la hidròlisi dels enllaços fosfodièster. Les nucleases que hidrolitzen nucleòtids dels extrems de les cadenes reben el nom d'exonucleases, mentre que les endonucleases fan talls dins de les cadenes. Les nucleases utilitzades més sovint en la biologia molecular són les endonucleases de restricció, que retallen l'ADN a seqüències específiques. Aquestes seqüències s'anomenen dianes de restricció, i solen ésser palindròmiques (es llegeixen igual en els dos sentits de la cadena d'ADN). Per exemple, l'enzim EcoRV mostrat a l'esquerra reconeix la seqüència de sis bases 5′-GAT|ATC-3′ i fa un tall a la línia vertical. A la natura, aquests enzims protegeixen els bacteris de la infecció per part de bacteriòfags, digerint l'ADN del fag quan penetra a la cèl·lula bacteriana, actuant com a part del sistema de restricció-modificació.[51] En la tecnologia, aquestes nucleases específiques per cada seqüència s'utilitzen en la clonació molecular i la identificació de l'ADN.
Els enzims anomenats ADN ligases poden tornar a ajuntar cadenes d'ADN tallades o trencades.[52] Les ligases són particularment importants en la replicació de l'ADN de la cadena retardada, car uneixen els segments curts d'ADN produïts a la forqueta de replicació en una còpia completa de la plantilla d'ADN. També es fan servir en la reparació de l'ADN i la recombinació genètica.[52]
Topoisomerases i helicases
Les topoisomerases són enzims amb activitat tant de nucleasa com de ligasa. Aquestes proteïnes canvien la quantitat de superenrotllament de l'ADN. Alguns d'aquests enzims funcionen tallant l'hèlix d'ADN i permetent la rotació d'una secció, reduint-ne així el nivell de superenrotllament; aleshores l'enzim segella el trencament de l'ADN.[53] Altres tipus d'aquests enzims són capaços de tallar una hèlix d'ADN i aleshores fer passar una segona cadena d'ADN per aquest trencament, reajuntant així l'hèlix.[54] Les topoisomerases són necessàries per molts processos relacionats amb l'ADN, com ara la replicació i la transcripció de l'ADN.[55]
Les helicases són proteïnes que són una mena de motor molecular. Utilitzen l'energia química dels nucleòsids trifosfats, majoritàriament ATP, per trencar els enllaços d'hidrogen entre les bases i desenrotllar la doble hèlix d'ADN en cadenes separades.[56] Aquests enzims són essencials per la majoria de processos en què enzims necessiten accedir a les bases de l'ADN.
Polimerases
Les polimerases són enzims que sintetitzen cadenes de polinucleòtids a partir de nucleòsids trifosfats. La seqüència dels seus productes són còpies de cadenes ja existents de polinucleòtids, anomenades «plantilles». Aquests enzims funcionen afegint nucleòtids al grup hidroxil 3′ del nucleòtid previ d'una cadena d'ADN. Per consegüent, totes les polimerases funcionen en direcció 5′ a 3′.[57] A la zona activa d'aquests enzims, el nucleòsid trifosfat entrant aparella les bases amb la plantilla: això permet a les polimerases sintetitzar amb precisió la cadena complementària de la seva plantilla. Les polimerases es classifiquen segons el tipus de plantilla que utilitzen.
En la replicació de l'ADN, una ADN polimerasa ADN dependent crea una còpia d'una seqüència d'ADN. La precisió és vital en aquest procés, de manera que moltes d'aquestes polimerases tenen una activitat de correcció de proves. Aquí, la polimerasa reconeix els errors ocasionals de la reacció de síntesi per la manca d'aparellament de bases entre els nucleòtids mal aparellats. Si es detecta un malaparellament, s'engega una activitat d'exonucleasa 3′ a 5′ i la base incorrecta és eliminada.[58] En la majoria d'organismes les ADN polimerases funcionen en un gran complex anomenat replisoma que conté múltiples subunitats accessòries, com ara l'abraçadora d'ADN o les helicases.[59]
Les ADN polimerases ARN dependents són una classe especialitzada de polimerases que copien la seqüència d'una cadena d'ARN en ADN. Inclouen la transcriptasa inversa, que és un enzim víric implicat en la infecció de les cèl·lules per retrovirus, o la telomerasa, que és necessària per a la replicació dels telòmers.[60][61] La telomerasa és una polimerasa inusual, car conté la seva pròpia plantilla d'ARN com a part de la seva estructura.[32]
La transcripció és executada per una ARN polimerasa ADN dependent que copia la seqüència d'una cadena d'ADN en ARN. Per començar a transcriure un gen, l'ARN polimerasa s'uneix a una seqüència d'ADN anomenada «promotor» i separa les cadenes d'ADN. Aleshores copia la seqüència gènica a un transcrit d'ARN missatger fins que arriba a una regió de l'ADN, el «terminador», on s'atura i se separa de l'ADN. Com en les ADN polimerases ADN dependents humanes, l'ARN polimerasa II, l'enzim que transcriu la majoria de gens del genoma humà, opera com a part d'un gran complex proteic amb múltiples subunitats reguladores i accessòries.[62]
Recombinació genètica

|

|

Una hèlix d'ADN no interacciona habitualment amb altres segments d'ADN, i en les cèl·lules humanes els diferents cromosomes fins i tot ocupen parts separades del nucli anomenades "territoris cromosòmics".[64] Aquesta separació física dels diferents cromosomes és important per la capacitat de l'ADN de funcionar com a dipòsit estable d'informació, car una de les poques ocasions en què interaccionen els cromosomes és durant l'encreuament cromosòmic quan es recombinen. L'encreuament cromosòmic és quan dues hèlixs d'ADN es trenquen, s'intercanvien una secció i aleshores es tornen a ajuntar.
La recombinació permet als cromosomes intercanviar informació genètica i produeix noves combinacions de gens, cosa que augmenta l'eficàcia de la selecció natural i pot ser important en l'evolució ràpida de noves proteïnes.[65] La recombinació genètica també pot estar implicada en la reparació de l'ADN, particularment en la resposta cel·lular als trencaments de la cadena doble.[66]
La forma més comuna d'encreuament cromosòmic és la recombinació homòloga, en què els dos cromosomes implicats tenen seqüències molt similars. La recombinació no homòloga pot danyar les cèl·lules, car pot produir translocacions cromosòmiques i anormalitats genètiques. La reacció de recombinació és catalitzada per enzims coneguts com a recombinases, com ara RAD51.[67] El primer pas de la recombinació és un trencament de la cadena doble causat o bé per una endonucleasa o bé per danys a l'ADN.[68] Aleshores, una sèrie de passos catalitzats en part per la recombinasa porta a la unió de les dues hèlixs a través d'almenys una junció de Holliday, en què un segment d'una sola cadena de cada hèlix és unida a la cadena complementària de l'altra hèlix. La junció de Holliday és una estructura de junció tetraèdrica que es pot moure al llarg del parell de cromosomes, canviant una cadena per una altra. Aleshores, la reacció de recombinació és aturada pel trencament de la junció i el relligament de l'ADN alliberat.[69]
Evolució
L'ADN conté la informació genètica que permet funcionar, créixer i reproduir-se a tots els éssers vius actuals. Tanmateix, no està clar durant quant de temps dels 4.000 milions d'anys d'història de la vida l'ADN ha jugat aquest paper, car s'ha proposat que les primeres formes de vida podrien haver utilitzat ARN com a material genètic.[70][71] L'ARN podria haver servit com a part central del metabolisme cel·lular primitiu, car és capaç tant de transmetre informació genètica com de catalitzar reaccions com a part dels ribozims.[72] Aquest antic món d'ARN en què aquest àcid nucleic hauria estat utilitzat tant per la catàlisi com per la genètica podria haver influït en l'evolució del codi genètic actual basat en quatre bases de nucleòtids. Això hauria passat perquè el nombre de bases úniques d'un organisme és un equilibri entre un nombre reduït de bases, que augmenta la precisió de la replicació, i un nombre gran de bases, que augmenta l'eficàcia catalitzadora dels ribozims.[73]
Desafortunadament, no hi ha proves directes dels sistemes genètics ancestrals, car resulta impossible recuperar ADN de la majoria de fòssils. Això es deu al fet que l'ADN sobreviu menys d'un milió d'anys al medi ambient i a poc a poc es degrada en petits fragments en dissolució.[74] S'ha al·legat el descobriment d'ADN més antic, notablement l'informe de l'aïllament d'un bacteri viable d'un cristall de sal de 250 milions d'anys d'antiguitat,[75] però aquestes al·legacions són controvertides.[76][77]
Usos en la tecnologia
Enginyeria genètica
S'han desenvolupat mètodes per purificar ADN dels organismes, com ara l'extracció amb fenol-cloroform, i manipular-lo al laboratori, com ara les digestions de restricció i la reacció en cadena de la polimerasa. La biologia i la bioquímica actuals fan un ús extens d'aquestes tècniques en la tecnologia de l'ADN recombinant. L'ADN recombinant és una seqüència d'ADN elaborada artificialment pels humans que ha estat assemblada a partir d'altres seqüències d'ADN. Es pot transformar en organismes en forma de plasmidis o en el format adequat, utilitzant un vector víric.[78] Els organismes genèticament modificats es poden utilitzar per generar productes com ara proteïnes recombinants, utilitzades en la investigació mèdica,[79] o ser cultivats en l'agricultura.[80][81]
Ciència forense
Els científics forenses poden utilitzar l'ADN de sang, semen, pell, saliva o pèl trobats a l'escena d'un crim per identificar l'ADN d'un individu, com ara el perpetrador del crim. Aquest procés rep el nom d'identificació genètica, o més precisament, anàlisi de perfils d'ADN. En l'anàlisi de perfils d'ADN, es compara la llargada de seccions variables d'ADN repetitiu de diferents persones, com ara seqüències repetitives en tàndem i minisatèl·lits. Aquest mètode és habitualment una tècnica extremament fiable per identificar l'ADN corresponent.[82] Tanmateix, la identificació es pot complicar si l'escena està contaminada amb ADN de diverses persones.[83] L'anàlisi de perfils d'ADN fou desenvolupada el 1984 pel genetista britànic Sir Alec Jeffreys,[84] i fou utilitzada per primer cop en la ciència forense el 1988, per condemnar Colin Pitchfork pels assassinats d'Enderby.[85]
Als condemnats per determinats tipus de crims se'ls pot requerir que donin una mostra d'ADN per posar-la en una base de dades. Això ha ajudat els investigadors a resoldre casos antics en què només s'havia obtingut una mostra d'ADN de l'escena. L'anàlisi de perfils d'ADN també es pot fer servir per identificar les víctimes en els accidents amb molts morts.[86] D'altra banda, molts condemnats han estat posats en llibertat basant-se en les tècniques d'ADN, que no estaven disponibles quan es produí el crim.
Bioinformàtica
La bioinformàtica concerneix la manipulació, la cerca i la mineria de dades de seqüències d'ADN. El desenvolupament de les tècniques per emmagatzemar i cercar en seqüències d'ADN ha conduït a avenços extensament aplicats en informàtica, especialment en algoritmes de recerca de cadenes, l'aprenentatge automàtic i la teoria de bases de dades.[87] Els algoritmes de recerca o aparellament de cadenes, que troben una ocurrència d'una seqüència de lletres dins d'una seqüència de lletres més gran, foren desenvolupats per buscar seqüències específiques de nucleòtids.[88] En altres aplicacions com ara processadors de text, sovint són suficients algoritmes simples per resoldre aquest problema, però les seqüències d'ADN fan que aquests algoritmes presentin un comportament de quasi-pitjor-cas a causa del seu nombre reduït de caràcters diferents. El problema relacionat de l'alineament de seqüències aspira a identificar seqüències homòlogues i ubicar les mutacions específiques que les fan diferents. Aquestes tècniques, especialment l'alineament múltiple de seqüències, són utilitzades en l'estudi de les relacions filogenètiques i el funcionament de les proteïnes[89] Els conjunts de dades que representen totes les seqüències d'ADN d'un genoma, com els que produeix el Projecte Genoma Humà, són difícils d'utilitzar sense marcacions, que etiqueten la situació dels gens i els elements reguladors de cada cromosoma. Les regions de les seqüències d'ADN que tenen els patrons específics associats amb els gens codificadors de proteïnes o ARN es poden identificar per mitjà d'algoritmes de detecció de gens, que permeten als investigadors predir la presència de productes gènics en un organisme fins i tot abans d'aïllar-los experimentalment.[90]
Nanotecnologia de l'ADN

La nanotecnologia de l'ADN utilitza les propietats úniques de reconeixement molecular de l'ADN i altres àcids nucleics per crear complexos d'ADN ramificats autoassemblants amb propietats útils.[91] Així doncs, l'ADN s'utilitza com a material estructural i no com a portador d'informació biològica. Això ha conduït a la creació de llibrets periòdics bidimensionals (tant basats en lloses com mitjançant el mètode de l'origami d'ADN), així com estructures tridimensionals amb forma de políedre.[92] També s'han demostrat dispositius nanomecànics i l'autoassemblatge algorítmic,[93] i aquestes estructures d'ADN s'han utilitzat com a plantilla per l'arranjament d'altres molècules com ara les nanopartícules d'or o les proteïnes estreptavidines.[94]
Història i antropologia
Com que l'ADN acumula mutacions al llarg del temps que després són heretades, conté informació històrica, i per mitjà de la comparació de les seqüències d'ADN els genetistes poden inferir la història evolutiva dels organismes, és a dir, la seva filogènesi.[95] Aquest camp de la filogènia és una eina potent en la biologia evolutiva. Si es comparen les seqüències d'ADN dins d'una espècie, els genetistes de poblacions poden esbrinar la història de poblacions concretes. Això es pot utilitzar en estudis, des de la genètica ecològica fins a l'antropologia; per exemple, s'estan utilitzant proves d'ADN per intentar identificar les deu tribus perdudes d'Israel.[96][97]
També s'ha utilitzat l'ADN per investigar les relacions familiars modernes, com quan es va determinar l'estreta relació familiar entre els descendents de Sally Hemings i Thomas Jefferson. Aquest ús està estretament relacionat amb l'ús de l'ADN en investigacions criminals, del qual s'ha parlat més amunt. De fet, algunes investigacions criminals s'han resolt quan ADN de l'escena del crim s'ha relacionat amb el de familiars de l'individu culpable.[98]
Història de l'estudi de l'ADN
L'ADN fou aïllat per primer cop pel metge suís Friedrich Miescher, que el 1869 descobrí una substància microscòpica al pus de benes quirúrgiques per llençar. Com que es trobava al nucli de les cèl·lules, l'anomenà «nucleïna».[99] El 1919, Phoebus Levene identificà els nucleòtids compostos d'una base nitrogenada, un sucre i fosfats.[100] Levene suggerí que l'ADN consistia en una cadena de nucleòtids enllaçats pels grups fosfat. Tanmateix, Levene creia que la cadena era curta i que les bases es repetien en un ordre fix. Val a dir que en aquells temps era àmpliament acceptat que la informació genètica residia en les proteïnes i no pas en els àcids nucleics, i que aquests últims tenien un paper estructural. El 1937, William Astbury produí els primers patrons de difracció de rajos X que demostraven que l'ADN té una estructura regular.[101]
El 1928, Frederick Griffith descobrí que els trets de la forma «llisa» de Pneumococcus es podien transferir a la forma «rugosa» del mateix bacteri si es mesclaven bacteris "llisos" morts amb la forma "rugosa" vivent.[102] Aquest sistema oferí el primer indici clar que l'ADN portava informació genètica — l'experiment d'Avery, MacLeod i McCarty — quan Oswald Avery, juntament amb els seus col·laboradors Colin MacLeod i Maclyn McCarty, identificà l'ADN com a principi transformador el 1943.[103] El paper de l'ADN en l'herència fou confirmat el 1952, quan Alfred Hershey i Martha Chase demostraren en l'experiment de Hershey i Chase que l'ADN és el material genètic del fag T2.[104]
-
Controvèrsia de l'hèlix d'ADN
El 1953, James D. Watson i Francis Crick suggeriren el qual actualment s'accepta com el primer model de doble hèlix correcte de l'estructura de l'ADN a la revista Nature.[4] El seu model molecular de doble hèlix de l'ADN es basava en una única imatge per difracció de rajos X (etiquetada com a "Foto 51")[105] presa per Rosalind Franklin i Raymond Gosling al maig del 1952, així com la informació que les bases d'ADN estaven aparellades - obtinguda a través de comunicacions privades amb Erwin Chargaff als anys anteriors. Les regles de Chargaff tingueren un paper molt important en la determinació de les configuracions de doble hèlix tant pel B-ADN com per l'A-ADN.
Les proves experimentals que donaven suport el model de Watson i Crick foren publicades en una sèrie de cinc articles al mateix número de Nature.[106] D'aquests, l'article de Franklin i Goslin fou la primera publicació de les seves pròpies dades de la difracció de rajos X, i del seu mètode analític original que recolzava en part el model de Watson i Crick;[107][108] aquest número també contenia un article sobre l'estructura de l'ADN de Maurice Wilkins i dos dels seus col·laboradors, les anàlisis i els patrons de rajos X del B-ADN in vivo dels quals també recolzaven la presència in vivo de les configuracions en doble hèlix de l'ADN, com ho proposaven Crick i Watson en el seu model molecular de doble hèlix de l'ADN a les dues pàgines anteriors de Nature.[109] El 1962, després de la mort de Franklin, Watson, Crick i Wilkins foren guardonats conjuntament amb el Premi Nobel de Fisiologia o Medicina.[110] Malauradament, les regles del Premi Nobel d'aquell temps només permetien guardonar guanyadors vivents, però encara dura un intens debat sobre a qui se li hauria d'atribuir aquest descobriment.[111]
En una presentació influent del 1957, Crick postulà el dogma central de la biologia molecular, que preveia la relació entre l'ADN, l'ARN i les proteïnes, i articulà la "hipòtesi de l'adaptador".[112] La confirmació final del mecanisme de replicació que implicava l'estructura de doble hèlix arribà el 1958 amb l'experiment de Meselson i Stahl.[113] Treballs posteriors de Crick i col·laboradors demostraren que el codi genètic es basava en triplets no solapats de bases, anomenats codons, permetent a Har Gobind Khorana, Robert W. Holley i Marshall Warren Nirenberg desxifrar el codi genètic.[114] Aquests descobriments representen el naixement de la genètica molecular.
Referències
- ↑ Saenger, Wolfram. Principles of Nucleic Acid Structure (en anglès). Nova York: Springer-Verlag, 1984. ISBN 0-387-90762-9.
- ↑ Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walters, Peter. Molecular Biology of the Cell; Fourth Edition (en anglès). Nova York i Londres: Garland Science, 2002. ISBN 0-8153-3218-1. OCLC 145080076.
- ↑ Irobalieva, Rossitza N.; Fogg, Jonathan M.; Catanese Jr, Daniel J.; Sutthibutpong, Thana; Chen, Muyuan; Barker, Anna K.; Ludtke, Steven J.; Harris, Sarah A.; Schmid, Michael F. «Structural diversity of supercoiled DNA» (en anglès). Nature Communications, 6, 12-10-2015, pàg. 8.440. DOI: 10.1038/ncomms9440. PMC: 4608029. PMID: 26455586.
- ↑ 4,0 4,1 Watson J.D. and Crick F.H.C. «A Structure for Deoxyribose Nucleic Acid» (PDF) (en anglès). Nature, 171, 1953, pàg. 737–738. DOI: 10.1038/171737a0. PMID: 13054692 [Consulta: 4 maig 2009].
- ↑ «The dimensions of DNA in solution» (en anglès). J Mol Biol, 152, 1, 1981, pàg. 153–61. DOI: 10.1016/0022-2836(81)90099-1. PMID: 7338906.
- ↑ Gregory S, et al. «The DNA sequence and biological annotation of human chromosome 1». Nature, 441, 7091, 2006, pàg. 315-21. DOI: 10.1038/nature04727. PMID: 16710414.
- ↑ Watson, J.; Crick, F. «Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid» (PDF). Nature, 171, 4356, 1953, pàg. 737–8. DOI: 10.1038/171737a0. PMID: 13054692.
- ↑ 8,0 8,1 8,2 Berg J.; Tymoczko J.; Stryer L. (2002) Biochemistry. W. H. Freeman and Company ISBN 0-7167-4955-6
- ↑ Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents IUPAC-IUB Commission on Biochemical Nomenclature (CBN), Consultat el 3 gener 2006
- ↑ Ghosh A, Bansal M «A glossary of DNA structures from A to Z». Acta Crystallogr D Biol Crystallogr, 59, Pt 4, 2003, pàg. 620–6. DOI: 10.1107/S0907444903003251. PMID: 12657780.
- ↑ Klose R, Bird A «Genomic DNA methylation: the mark and its mediators». Trends Biochem Sci, 31, 2, 2006, pàg. 89–97. DOI: 10.1016/j.tibs.2005.12.008. PMID: 16403636.
- ↑ Bird A «DNA methylation patterns and epigenetic memory». Genes Dev, 16, 1, 2002, pàg. 6–21. DOI: 10.1101/gad.947102. PMID: 11782440.
- ↑ Walsh C, Xu G «Cytosine methylation and DNA repair». Curr Top Microbiol Immunol, 301, 2006, pàg. 283–315. DOI: 10.1007/3-540-31390-7_11. PMID: 16570853.
- ↑ Kriaucionis S, Heintz N «The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain». Science, 324, 5929, Maig 2009, pàg. 929–30. DOI: 10.1126/science.1169786. PMID: 19372393.
- ↑ Ratel D, Ravanat J, Berger F, Wion D «N6-methyladenine: the other methylated base of DNA». Bioessays, 28, 3, 2006, pàg. 309–15. DOI: 10.1002/bies.20342. PMID: 16479578.
- ↑ Gommers-Ampt J, Van Leeuwen F, de Beer A, Vliegenthart J, Dizdaroglu M, Kowalak J, Crain P, Borst P «beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei». Cell, 75, 6, 1993, pàg. 1129–36. DOI: 10.1016/0092-8674(93)90322-H. PMID: 8261512.
- ↑ Creat a partir de PDB 1JDG Arxivat 2008-09-22 a Wayback Machine.
- ↑ Douki T, Reynaud-Angelin A, Cadet J, Sage E «Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation». Biochemistry, 42, 30, 2003, pàg. 9221–6. DOI: 10.1021/bi034593c. PMID: 12885257.
- ↑ Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S «Hydroxyl radicals and DNA base damage». Mutat Res, 424, 1-2, 1999, pàg. 9–21. PMID: 10064846.
- ↑ Beckman KB, Ames BN «Oxidative decay of DNA». J. Biol. Chem., 272, 32, Agost 1997, pàg. 19633–6. PMID: 9289489.[Enllaç no actiu]
- ↑ Valerie K, Povirk L «Regulation and mechanisms of mammalian double-strand break repair». Oncogene, 22, 37, 2003, pàg. 5792–812. DOI: 10.1038/sj.onc.1206679. PMID: 12947387.
- ↑ Ferguson L, Denny W «The genetic toxicology of acridines». Mutat Res, 258, 2, 1991, pàg. 123–60. PMID: 1881402.
- ↑ Jeffrey A «DNA modification by chemical carcinogens». Pharmacol Ther, 28, 2, 1985, pàg. 237–72. DOI: 10.1016/0163-7258(85)90013-0. PMID: 3936066.
- ↑ Stephens T, Bunde C, Fillmore B «Mechanism of action in thalidomide teratogenesis». Biochem Pharmacol, 59, 12, 2000, pàg. 1489–99. DOI: 10.1016/S0006-2952(99)00388-3. PMID: 10799645.
- ↑ Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A «Intercalators as anticancer drugs». Curr Pharm Des, 7, 17, 2001, pàg. 1745–80. DOI: 10.2174/1381612013397113. PMID: 11562309.
- ↑ Venter J, et al. «The sequence of the human genome». Science, 291, 5507, 2001, pàg. 1304–51. DOI: 10.1126/science.1058040. PMID: 11181995.
- ↑ Thanbichler M, Wang S, Shapiro L «The bacterial nucleoid: a highly organized and dynamic structure» (en anglès). J Cell Biochem, 96, 3, 2005, pàg. 506–21. DOI: 10.1002/jcb.20519. PMID: 15988757.
- ↑ Wolfsberg T, McEntyre J, Schuler G «Guide to the draft human genome». Nature, 409, 6822, 2001, pàg. 824–6. DOI: 10.1038/35057000. PMID: 11236998.
- ↑ Gregory T «The C-value enigma in plants and animals: a review of parallels and an appeal for partnership». Ann Bot (Lond), 95, 1, 2005, pàg. 133–46. DOI: 10.1093/aob/mci009. PMID: 15596463.
- ↑ The ENCODE Project Consortium «Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project». Nature, 447, 7146, 2007, pàg. 799–816. DOI: 10.1038/nature05874.
- ↑ Creat a partir de PDB 1MSW
- ↑ 32,0 32,1 Nugent C.; Lundblad V. «The telomerase reverse transcriptase: components and regulation». Genes Dev, 12, 8, 1998, pàg. 1073–85. DOI: 10.1101/gad.12.8.1073. PMID: 9553037.
- ↑ Pidoux A, Allshire R «The role of heterochromatin in centromere function». Philos Trans R Soc Lond B Biol Sci, 360, 1455, 2005, pàg. 569–79. DOI: 10.1098/rstb.2004.1611. PMC: 1569473. PMID: 15905142.
- ↑ Harrison P, Hegyi H, Balasubramanian S, Luscombe N, Bertone P, Echols N, Johnson T, Gerstein M «Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22». Genome Res, 12, 2, 2002, pàg. 272–80. DOI: 10.1101/gr.207102. PMID: 11827946.
- ↑ Harrison P, Gerstein M «Studying genomes through the aeons: protein families, pseudogenes and proteome evolution». J Mol Biol, 318, 5, 2002, pàg. 1155–74. DOI: 10.1016/S0022-2836(02)00109-2. PMID: 12083509.
- ↑ Albà M «Replicative DNA polymerases». Genome Biol, 2, 1, 2001, pàg. REVIEWS3002. DOI: 10.1186/gb-2001-2-1-reviews3002. PMC: 150442. PMID: 11178285.[Enllaç no actiu]
- ↑ Sandman K, Pereira S, Reeve J «Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome». Cell Mol Life Sci, 54, 12, 1998, pàg. 1350–64. DOI: 10.1007/s000180050259. PMID: 9893710.
- ↑ Dame RT «The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin». Mol. Microbiol., 56, 4, 2005, pàg. 858–70. DOI: 10.1111/j.1365-2958.2005.04598.x. PMID: 15853876.
- ↑ Luger K, Mäder A, Richmond R, Sargent D, Richmond T «Crystal structure of the nucleosome core particle at 2.8 A resolution». Nature, 389, 6648, 1997, pàg. 251–60. DOI: 10.1038/38444. PMID: 9305837.
- ↑ Jenuwein T, Allis C «Translating the histone code». Science, 293, 5532, 2001, pàg. 1074–80. DOI: 10.1126/science.1063127. PMID: 11498575.
- ↑ Ito T «Nucleosome assembly and remodelling». Curr Top Microbiol Immunol, 274, pàg. 1-22. PMID: 12596902.
- ↑ Thomas J «HMG1 and 2: architectural DNA-binding proteins». Biochem Soc Trans, 29, Pt 4, 2001, pàg. 395–401. DOI: 10.1042/BST0290395. PMID: 11497996.
- ↑ Grosschedl R, Giese K, pàg.l J «HMG domain proteins: architectural elements in the assembly of nucleoprotein structures». Trends Genet, 10, 3, 1994, pàg. 94–100. DOI: 10.1016/0168-9525(94)90232-1. PMID: 8178371.
- ↑ Iftode C, Daniely Y, Borowiec J «Replication protein A (RPA): the eukaryotic SSB». Crit Rev Biochem Mol Biol, 34, 3, 1999, pàg. 141–80. DOI: 10.1080/10409239991209255. PMID: 10473346.
- ↑ Creat a partir de PDB 1LMB
- ↑ Myers L, Kornberg R «Mediator of transcriptional regulation». Annu Rev Biochem, 69, 2000, pàg. 729–49. DOI: 10.1146/annurev.biochem.69.1.729. PMID: 10966474.
- ↑ Spiegelman B, Heinrich R «Biological control through regulated transcriptional coactivators». Cell, 119, 2, 2004, pàg. 157–67. DOI: 10.1016/j.cell.2004.09.037. PMID: 15479634.
- ↑ Li Z, Van Calcar S, Qu C, Cavenee W, Zhang M, Ren B «A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells». Proc Natl Acad Sci USA, 100, 14, 2003, pàg. 8164–9. DOI: 10.1073/pnas.1332764100. PMC: 166200. PMID: 12808131.
- ↑ Pabo C, Sauer R «Protein-DNA recognition». Annu Rev Biochem, 53, 1984, pàg. 293–321. DOI: 10.1146/annurev.bi.53.070184.001453. PMID: 6236744.
- ↑ Creat a partir de PDB 1RVA
- ↑ Bickle T, Krüger D «Biology of DNA restriction». Microbiol Rev, 57, 2, 1993, pàg. 434–50. PMC: 372918. PMID: 8336674.
- ↑ 52,0 52,1 Doherty A, Suh S «Structural and mechanistic conservation in DNA ligases». Nucleic Acids Res, 28, 21, 2000, pàg. 4051–8. DOI: 10.1093/nar/28.21.4051. PMC: 113121. PMID: 11058099.
- ↑ Champoux J. «DNA topoisomerases: structure, function, and mechanism». Annu Rev Biochem, 70, 2001, pàg. 369–413. DOI: 10.1146/annurev.biochem.70.1.369. PMID: 11395412.
- ↑ Schoeffler A, Berger J «Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism». Biochem Soc Trans, 33, Pt 6, 2005, pàg. 1465–70. DOI: 10.1042/BST20051465. PMID: 16246147.
- ↑ Wang J. «Cellular roles of DNA topoisomerases: a molecular perspective». Nat Rev Mol Cell Biol, 3, 6, 2002, pàg. 430–40. DOI: 10.1038/nrm831. PMID: 12042765.
- ↑ Tuteja N, Tuteja R «Unraveling DNA helicases. Motif, structure, mechanism and function». Eur J Biochem, 271, 10, 2004, pàg. 1849–63. DOI: 10.1111/j.1432-1033.2004.04094.x. PMID: 15128295.
- ↑ Joyce C, Steitz T «Polymerase structures and function: variations on a theme?». J Bacteriol, 177, 22, 1995, pàg. 6321–9. PMC: 177480. PMID: 7592405.
- ↑ Hubscher U, Maga G, Spadari S «Eukaryotic DNA polymerases». Annu Rev Biochem, 71, 2002, pàg. 133–63. DOI: 10.1146/annurev.biochem.71.090501.150041. PMID: 12045093.
- ↑ Johnson A, O'Donnell M «Cellular DNA replicases: components and dynamics at the replication fork». Annu Rev Biochem, 74, 2005, pàg. 283–315. DOI: 10.1146/annurev.biochem.73.011303.073859. PMID: 15952889.
- ↑ Greider C.; Blackburn E. «Identification of a specific telomere terminal transferase activity in Tetrahymena extracts». Cell, 43, 2 Pt 1, 1985, pàg. 405–13. DOI: 10.1016/0092-8674(85)90170-9. PMID: 3907856.
- ↑ Tarrago-Litvak L, Andréola M, Nevinsky G, Sarih-Cottin L, Litvak S «The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention». Faseb J, 8, 8, Maig 1994, pàg. 497–503. PMID: 7514143.
- ↑ Martinez E «Multi-protein complexes in eukaryotic gene transcription». Plant Mol Biol, 50, 6, 2002, pàg. 925–47. DOI: 10.1023/A:1021258713850. PMID: 12516863.
- ↑ Creat a partir de PDB 1M6G
- ↑ Cremer T, Cremer C «Chromosome territories, nuclear architecture and gene regulation in mammalian cells». Nat Rev Genet, 2, 4, 2001, pàg. 292–301. DOI: 10.1038/35066075. PMID: 11283701.
- ↑ Pál C, Papp B, Lercher M «An integrated view of protein evolution». Nat Rev Genet, 7, 5, 2006, pàg. 337–48. DOI: 10.1038/nrg1838. PMID: 16619049.
- ↑ O'Driscoll M, Jeggo P «The role of double-strand break repair - insights from human genetics». Nat Rev Genet, 7, 1, 2006, pàg. 45–54. DOI: 10.1038/nrg1746. PMID: 16369571.
- ↑ Vispé S, Defais M «Mammalian Rad51 protein: a RecA homologue with pleiotropic functions». Biochimie, 79, 9-10, 1997, pàg. 587–92. DOI: 10.1016/S0300-9084(97)82007-X. PMID: 9466696.
- ↑ Neale MJ, Keeney S «Clarifying the mechanics of DNA strand exchange in meiotic recombination». Nature, 442, 7099, 2006, pàg. 153–8. DOI: 10.1038/nature04885. PMID: 16838012.
- ↑ Dickman M, Ingleston S, Sedelnikova S, Rafferty J, Lloyd R, Grasby J, Hornby D «The RuvABC resolvasome». Eur J Biochem, 269, 22, 2002, pàg. 5492–501. DOI: 10.1046/j.1432-1033.2002.03250.x. PMID: 12423347.
- ↑ Joyce G «The antiquity of RNA-based evolution». Nature, 418, 6894, 2002, pàg. 214–21. DOI: 10.1038/418214a. PMID: 12110897.
- ↑ Orgel L «Prebiotic chemistry and the origin of the RNA world» (PDF). Crit Rev Biochem Mol Biol, 39, 2, 2004, pàg. 99–123. DOI: 10.1080/10409230490460765. PMID: 15217990.
- ↑ Davenport R «Ribozymes. Making copies in the RNA world». Science, 292, 5520, 2001, pàg. 1278. DOI: 10.1126/science.292.5520.1278a. PMID: 11360970.
- ↑ Szathmáry E «What is the optimum size for the genetic alphabet?» (PDF). Proc Natl Acad Sci USA, 89, 7, 1992, pàg. 2614–8. Arxivat de l'original el 2008-06-25. DOI: 10.1073/pnas.89.7.2614. PMID: 1372984 [Consulta: 23 juliol 2009].
- ↑ Lindahl T «Instability and decay of the primary structure of DNA». Nature, 362, 6422, 1993, pàg. 709–15. DOI: 10.1038/362709a0. PMID: 8469282.
- ↑ Vreeland R, Rosenzweig W, Powers D «Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal». Nature, 407, 6806, 2000, pàg. 897–900. DOI: 10.1038/35038060. PMID: 11057666.
- ↑ Hebsgaard M, Phillips M, Willerslev E «Geologically ancient DNA: fact or artefact?». Trends Microbiol, 13, 5, 2005, pàg. 212–20. DOI: 10.1016/j.tim.2005.03.010. PMID: 15866038.
- ↑ Nickle D, Learn G, Rain M, Mullins J, Mittler J «Curiously modern DNA for a "250 million-year-old" bacterium». J Mol Evol, 54, 1, 2002, pàg. 134–7. DOI: 10.1007/s00239-001-0025-x. PMID: 11734907.
- ↑ Goff SP, Berg P «Construction of hybrid viruses containing SV40 and lambda phage DNA segments and their propagation in cultured monkey cells». Cell, 9, 4 PT 2, 1976, pàg. 695–705. DOI: 10.1016/0092-8674(76)90133-1. PMID: 189942.
- ↑ Houdebine L «Transgenic animal models in biomedical research». Methods Mol Biol, 360, pàg. 163–202. PMID: 17172731.
- ↑ Daniell H, Dhingra A «Multigene engineering: dawn of an exciting new era in biotechnology». Curr Opin Biotechnol, 13, 2, 2002, pàg. 136–41. DOI: 10.1016/S0958-1669(02)00297-5. PMID: 11950565.
- ↑ Job D «Plant biotechnology in agriculture». Biochimie, 84, 11, 2002, pàg. 1105–10. DOI: 10.1016/S0300-9084(02)00013-5. PMID: 12595138.
- ↑ Collins A, Morton N «Likelihood ratios for DNA identification» (PDF). Proc Natl Acad Sci USA, 91, 13, 1994, pàg. 6007–11. DOI: 10.1073/pnas.91.13.6007. PMID: 8016106.
- ↑ Weir B, Triggs C, Starling L, Stowell L, Walsh K, Buckleton J «Interpreting DNA mixtures». J Forensic Sci, 42, 2, 1997, pàg. 213–22. PMID: 9068179.
- ↑ Jeffreys A, Wilson V, Thein S «Individual-specific 'fingerprints' of human DNA». Nature, 316, 6023, 1985, pàg. 76–9. DOI: 10.1038/316076a0. PMID: 2989708.
- ↑ Colin Pitchfork — first murder conviction on DNA evidence also clears the prime suspect Arxivat 2006-12-14 a Wayback Machine. Forensic Science Service. Consultat el 23 desembre 2006
- ↑ «DNA Identification in Mass Fatality Incidents». National Institute of Justice, Setembre 2006. Arxivat de l'original el 2006-11-12. [Consulta: 23 juliol 2009].
- ↑ Baldi, Pierre; Brunak, Soren. Bioinformatics: The Machine Learning Approach. MIT Press, 2001. ISBN 978-0-262-02506-5. OCLC 45951728 57562233.
- ↑ Gusfield, Dan. Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology. Cambridge University Press, 15 gener 1997. ISBN 978-0-521-58519-4
- ↑ Sjölander K «Phylogenomic inference of protein molecular function: advances and challenges». Bioinformatics, 20, 2, 2004, pàg. 170–9. DOI: 10.1093/bioinformatics/bth021. PMID: 14734307.
- ↑ Mount DM. Bioinformatics: Sequence and Genome Analysis. 2. Cold Spring Harbor (Nova York): Cold Spring Harbor Laboratory Press, 2004. ISBN 0879697121. OCLC 55106399.
- ↑ Rothemund PW «Folding DNA to create nanoscale shapes and patterns». Nature, 440, 7082, Març 2006, pàg. 297–302. DOI: 10.1038/nature04586. PMID: 16541064.
- ↑ Andersen ES, Dong M, Nielsen MM, et al. «Self-assembly of a nanoscale DNA box with a controllable lid». Nature, 459, 7243, Maig 2009, pàg. 73–6. DOI: 10.1038/nature07971. PMID: 19424153.
- ↑ Ishitsuka Y, Ha T «DNA nanotechnology: a nanomachine goes live». Nat Nanotechnol, 4, 5, Maig 2009, pàg. 281-2. DOI: 10.1038/nnano.2009.101. PMID: 19421208.
- ↑ Aldaye FA, Palmer AL, Sleiman HF «Assembling materials with DNA as the guide». Science, 321, 5897, Setembre 2008, pàg. 1795–9. DOI: 10.1126/science.1154533. PMID: 18818351.
- ↑ Wray G «Dating branches on the tree of life using DNA». Genome Biol, 3, 1, 2002, pàg. REVIEWS0001. DOI: 10.1046/j.1525-142X.1999.99010.x. PMC: 150454. PMID: 11806830.[Enllaç no actiu]
- ↑ Lost Tribes of Israel, NOVA, Data d'emissió a PBS: 22 febrer 2000. Transcrit disponible a PBS.org, (consultat el 4 març 2006)
- ↑ Kleiman, Yaakov. "The Cohanim/DNA Connection: The fascinating story of how DNA studies confirm an ancient biblical tradition". Arxivat 2016-04-25 a Wayback Machine. aish.com (13 gener 2000). Consultat el 4 març 2006.
- ↑ Bhattacharya, Shaoni. "Killer convicted thanks to relative's DNA". newscientist.com (20 abril 2004). Consultat el 22 desembre 2006
- ↑ Dahm R «Discovering DNA: Friedrich Miescher and the early years of nucleic acid research». Hum. Genet., 122, 6, Gener 2008, pàg. 565–81. DOI: 10.1007/s00439-007-0433-0. PMID: 17901982.
- ↑ Levene P, «The structure of yeast nucleic acid». J Biol Chem, 40, 2, Desembre 1919, pàg. 415-24. Arxivat de l'original el 2009-06-29 [Consulta: 23 juliol 2009].
- ↑ Astbury W, «Nucleic acid». Symp. SOC. Exp. Bbl, 1, 66, 1947.
- ↑ Lorenz MG, Wackernagel W «Bacterial gene transfer by natural genetic transformation in the environment». Microbiol. Rev., 58, 3, Setembre 1994, pàg. 563–602. PMC: 372978. PMID: 7968924.
- ↑ Avery O, MacLeod C, McCarty M «Studies on the chemical nature of the substance inducing transformation of pneumococcal types. Inductions of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III». J Exp Med, 79, 2, 1944, pàg. 137–158. DOI: 10.1084/jem.79.2.137.
- ↑ Hershey A, Chase M «Independent functions of viral protein and nucleic acid in growth of bacteriophage» (PDF). J Gen Physiol, 36, 1, 1952, pàg. 39–56. DOI: 10.1085/jgp.36.1.39. PMID: 12981234.
- ↑ El patró de rajos X del B-ADN a la dreta d'aquesta imatge enllaçada fou obtingut per Rosalind Franklin i Raymond Gosling al maig del 1952 a nivells d'alta hidratació de l'ADN, i fou etiquetat com a "Foto 51"
- ↑ Nature Archives Double Helix of DNA: 50 Years
- ↑ Franklin, Rosalind and Gosling, Raymond «Molecular Configuration in Sodium Thymonucleate. Franklin R. and Gosling R.G» (
 PDF). Nature, 171, 1953, pàg. 740–1. DOI: 10.1038/171740a0. PMID: 13054694.
PDF). Nature, 171, 1953, pàg. 740–1. DOI: 10.1038/171740a0. PMID: 13054694.
- ↑ Imatge original per difracció de rajos X
- ↑ Wilkins M.H.F.; A.R. Stokes A.R. & Wilson, H.R. «Molecular Structure of Deoxypentose Nucleic Acids» (PDF). Nature, 171, 1953, pàg. 738–740. DOI: 10.1038/171738a0. PMID: 13054693.
- ↑ El Premi Nobel de Fisiologia o Medicina del 1962 Nobelprize.org Consultat el 22 desembre 2006
- ↑ Brenda Maddox. The double helix and the 'wronged heroine' (
 PDF). 421, 23 gener 2003, p. 407–408. DOI 10.1038/nature01399.
PDF). 421, 23 gener 2003, p. 407–408. DOI 10.1038/nature01399.
- ↑ Crick, F.H.C. On degenerate templates and the adaptor hypothesis. Arxivat 2008-10-01 a Wayback Machine. genome.wellcome.ac.uk (conferència, 1955). Consultat el 22 desembre 2006
 PDF
PDF
- ↑ Meselson M, Stahl F «The replication of DNA in Escherichia coli». PNAS, 44, 7, 1958, pàg. 671–82. DOI: 10.1073/pnas.44.7.671. PMID: 16590258.
- ↑ El Premi Nobel de Fisiologia o Medicina del 1968 Nobelprize.org Consultat el 22 desembre 2006
Bibliografia
- Calladine, Chris R.; Drew, Horace R.; Luisi, Ben F.; Travers, Andrew A.. Understanding DNA: the molecule & how it works. Amsterdam: Elsevier Academic Press, 2003. ISBN 0-12-155089-3.
- Dennis, Carina; Julie Clayton. 50 years of DNA. Basingstoke: Palgrave Macmillan, 2003. ISBN 1-4039-1479-6.
- Judson, Horace Freeland. The eighth day of creation: makers of the revolution in biology. Plainview (Nova York): CSHL Press, 1996. ISBN 0-87969-478-5.
- Olby, Robert C.. The path to the double helix: the discovery of DNA. Nova York: Dover Publications, 1994. ISBN 0-486-68117-3., publicat inicialment a l'octubre del 1974 per MacMillan, amb un prefaci de Francis Crick; llibre de text sobre l'ADN, revisat el 1994 amb una nota final de nou pàgines.
- Olby, Robert C.. Francis Crick: A Biography. Plainview (Nova York): Cold Spring Harbor Laboratory Press, 2009. ISBN 0-87969-798-9.
- Ridley, Matt. Francis Crick: discoverer of the genetic code. Ashland (Ohio): Eminent Lives, Atlas Books, 2006. ISBN 0-06-082333-X.
- Berry, Andrew; Watson, James D.. DNA: the secret of life. Nova York: Alfred A. Knopf, 2003. ISBN 0-375-41546-7.
- Stent, Gunther Siegmund; Watson, James D.. The double helix: a personal account of the discovery of the structure of DNA. Nova York: Norton, 1980. ISBN 0-393-95075-1.
- Wilkins, Maurice. The third man of the double helix the autobiography of Maurice Wilkins. Cambridge (Regne Unit): University Press, 2003. ISBN 0-19-860665-6.
Enllaços externs
- L'ADN al microscopi electrònic (anglès)
- Francis Crick and James Watson parlen a la BBC el 1962, 1972 i 1974(en anglès) Arxivat 2006-02-15 a Wayback Machine.
- Les contribucions de Rosalind Franklin a l'estudi de l'ADN(en anglès) Arxivat 2006-09-06 a Wayback Machine.
| Principals famíles bioquímiques | ||
| Àcids nucleics | Alcaloides | Aminoàcids | Carbohidrats | Carotenoides | Cofactors enzimàtics | Esteroides | Flavonoides | Glicòsids | Lípids | Pèptids | Policètids | Tetrapirrols | Terpens | ||
| Anàlegs d'àcids nucleics: | Tipus d'àcids nucleics | Anàlegs d'àcids nucleics : |
| Bases nitrogenades: | Adenina | Timina | Uracil | Guanina | Citosina | Purina | Pirimidina | |
|---|---|---|
| Nucleòsids: | Adenosina | Uridina | Guanosina | Citidina | Desoxiadenosina | Timidina | Desoxiguanosina | Desoxicitidina | |
| Nucleòtids: | AMP | UMP | GMP | CMP | ADP | UDP | GDP | CDP | ATP | UTP | GTP | CTP | AMPc | GMPc | ADPRc | |
| Desoxinucleòtids: | dAMP | TMP | dGMP | dCMP | dADP | TDP | dGDP | dCDP | dATP | TTP | dGTP | dCTP | |
| Àcids ribonucleics: | ARNm | ARNt | ARNr | ARNn | ARNnc | ARNmi | |
| Àcids desoxiribonucleics: | ADMmt | ADNc | |
| Anàlegs d'àcids nucleics: | AGN | APN | ATN | Morfolí | ARNin | |
| Seqüències: | Plasmidi | Còsmid | CAB | CAH | Cromosoma | Oligonucleòtid | |





