Genoma humà
| Per a altres significats, vegeu «Projecte Genoma Humà». |
El genoma humà és el genoma d'Homo sapiens, és a dir, les seqüències que contenen els 23 parells de cromosomes en el nucli de cada cèl·lula humana diploide. Dels 23 parells, 22 són cromosomes autosòmics i un parell determinant del sexe (dos cromosomes X en dones i un X i un Y en homes). Tenen una mida total aproximada de 3.200 milions de parells de bases d'ADN (3.200 Mb) que contenen uns 20.000 - 25.000 gens.[1] De les 3.200 Mb unes 2.950 Mb corresponen a eucromatina i unes 250 Mb a heterocromatina. El Projecte Genoma Humà va produir una seqüència de referència del genoma humà eucromàtic, utilitzat a tot el món en les ciències biomèdiques.
Les seqüències d'ADN que conforma el genoma humà contenen codificada la informació necessària per a l'expressió, altament coordinada i adaptable a l'ambient, del proteoma humà, és a dir, del conjunt de proteïnes de l'ésser humà. Les proteïnes, i no l'ADN, són les biomolècules efectores; organitzant-se en enormes xarxes funcionals d'interaccions. En definitiva, el proteoma fonamenta la particular morfologia i funcionalitat de cada cèl·lula. Així mateix, l'organització estructural i funcional de les diferents cèl·lules conforma cada teixit i cada òrgan i, finalment l'organisme viu en el seu conjunt. Així, el genoma humà conté la informació necessària per al desenvolupament bàsic d'un ésser humà complet.
El genoma humà presenta una densitat de gens molt inferior a la que inicialment s'havia predit, amb només un 1,5%[2] de la seva longitud constituïda per exons codificants de proteïnes. Un 70% està constiuit per ADN extragènic i un 30% per seqüències relacionades amb gens. Del total d'ADN extragènic, aproximadament un 70% correspon a repeticions disperses, de manera que, més o menys, la meitat del genoma humà correspon a seqüències repetitives d'ADN. D'altra banda, del total d'ADN relacionat amb els gens s'estima que el 95% correspon a ADN no codificant: pseudogèns, fragments de gens, introns, seqüències UTR, etc.
| Espècie | Mida del genoma (Mb) |
Nombre de gens |
|---|---|---|
| Mycoplasma genitalium | 0,58 | 500 |
| Streptococcus pneumoniae | 2,2 | 2300 |
| Escherichia coli | 4,6 | 4.400 |
| Saccharomyces cerevisiae | 12 | 5.800 |
| Caenorhabditis elegans | 97 | 19.000 |
| Arabidopsis thaliana | 125 | 25.500 |
| Drosophila melanogaster (mosca) | 180 | 13.700 |
| Oryza sativa (arròs) | 466 | 45-55.000 |
| Mus musculus (ratolí) | 2500 | 29.000 |
| Homo sapiens (ésser humà) | 3200 | 20.000 |
Components
[modifica]Cromosomes
[modifica]El genoma humà (com el de qualsevol organisme eucariota) està format per cromosomes, que són llargues seqüències contínues d'ADN altament organitzades espacialment (amb ajuda de proteïnes histones i no histones) per a adoptar una forma ultracondensada en metafase. Són observables amb microscòpia òptica convencional o de fluorescència mitjançant tècniques de citogenètica i s'ordenen formant un cariotip.
El cariotip humà conté un total de 24 cromosomes distints: 22 parells d'autosomes més 2 cromosomes sexuals que determinen el sexe de l'individu. Els cromosomes 1-22 van ser numerats en ordre decreixent de grandària sobre la base del cariotip. No obstant això, posteriorment va poder comprovar-se que el cromosoma 22 és en realitat major que el 21.

Les cèl·lules somàtiques d'un organisme posseïxen en el seu nucli un total de 46 cromosomes (23 parells): una dotació de 22 autosomes procedents de cada progenitor i un parell de cromosomes sexuals, un cromosoma X de la mare i un X o un Y del pare. (Vegeu imatge 1). Els gàmetes -òvuls i espermatozoides- posseeixen una dotació haploide de 23 cromosomes.
ADN intragènic
[modifica]Gens
[modifica]Un gen és la unitat bàsica de l'herència, i porta la informació genètica necessària per a la síntesi d'una proteïna (gens codificants) o d'un ARN no codificant (gens d'ARN). Està format per una seqüència promotora, que regula la seva expressió, i una seqüència que es transcriu, composta al seu torn per: seqüències UTR (regions flanquejants no traduïdes), necessàries per a la traducció i l'estabilitat de l'ARNm, exons (codificants) i introns, que són seqüències d'ADN no traduïdes situades entre dos exons que seran eliminades en el processament de l'ARNm (empalmament, splicing en anglès).
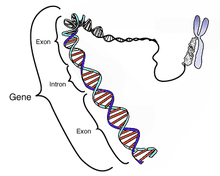
Actualment s'estima que el genoma humà conté entre 20.000 i 25.000 gens codificants de proteïnes, estimació molt inferior a les prediccions inicials que parlaven d'uns 100.000 gens o més. Això implica que el genoma humà té menys del doble de gens que organismes eucariotes molt més simples, com la mosca de la fruita o el nematode Caenorhabditis elegans. No obstant això, les cèl·lules humanes recorren àmpliament al splicing (empalmament) alternatiu per a produir diverses proteïnes diferents a partir d'un mateix gen, per la qual cosa el proteoma humà és més ample que el d'altres organismes molt més simples. En la pràctica, el genoma tan sols porta la informació necessària per a una expressió perfectament coordinada i regulada del conjunt de proteïnes que conformen el proteoma, sent aquest l'encarregat d'executar la major part de les funcions cel·lulars.
Gens d'ARN
[modifica]A més dels gens codificants de proteïnes, el genoma humà conté diversos milers de gens d'ARN, la transcripció dels quals produeix ARN de transferència (ARNt), ARN ribosòmic (ARNr), microARN (miARN), o altres gens ARN no codificants. Els ARN ribosòmics i de transferència són essencials en la constitució dels ribosomes i en la traducció de les proteïnes. Per la seva banda, els microARN tenen gran importància en la regulació de l'expressió gènica, estimant-se que fins a un 20-30% dels gens del genoma humà pot estar regulat pel mecanisme d'interferència per miARN. Fins al moment s'han identificat més de 300 gens de miARN i s'estima que en poden existir uns 500.
Distribució de gens
[modifica]A continuació es mostren alguns valors terme mitjà del genoma humà. Cap advertir, no obstant això, que l'enorme heterogeneïtat que presenten aquestes variables fa poc representatius als valors terme mitjà, encara que tenen valor orientatiu.
La densitat mitjana de gens és d'1 gen cada 100 kb, amb una grandària mitjana de 20-30 kb, i un nombre d'exons terme mitjà de 7-8 per cada gen, amb una grandària mitjana de 150 nucleòtids. La grandària mitjana d'un ARNm és d'1,8-2,2 kb, incloent les regions UTR (regions no traduïdes flanquejants), sent la longitud mitjana de la regió codificant d'1,4 kb.

El genoma humà es caracteritza per presentar una gran heterogeneïtat en la seva seqüència. En particular, la riquesa en bases de guanina (G) i citosina (C) enfront de les d'adenina (A) i timina (T) es distribuïx heterogèniament, amb regions molt riques en G+C flanquejades per regions molt pobres, sent el contingut mitjà de G+C del 41%, menor al teòricament esperat (50%). Aquesta heterogeneïtat està correlacionada amb la riquesa en gens, de manera que els gens tendeixen a concentrar-se en les regions més riques en G+C. Aquest fet era conegut ja des de fa anys gràcies a la separació mitjançant centrifugació en gradient de densitat de regions riques en G+C (que van rebre el nom d'isocors H; de l'anglès High) i regions riques en A+T (isocors L; de l'anglès Low).
Seqüències reguladores
[modifica]El genoma humà té diversos sistemes de regulació de l'expressió gènica, basats en la regulació de la unió de factors de transcripció a les seqüències promotores, en mecanismes de modificació epigenètica (metilació de l'ADN o metilació-acetilació d'histones) o en el control de l'accessibilitat als promotors determinada pel grau de condensació de la cromatina; tots ells molt interrelacionats. A més, hi ha altres sistemes de regulació a nivell del processament, estabilitat i traducció de l'ARNm, entre d'altres. Per tant, l'expressió gènica està intensament regulada, la qual cosa permet desenvolupar els múltiples fenotips que caracteritzen els diferents tipus cel·lulars d'un organisme eucariota multicel·lular, al mateix temps que dota a la cèl·lula de la plasticitat necessària per a adaptar-se a un mitjà canviant. No obstant això, tota la informació necessària per a la regulació de l'expressió gènica, en funció de l'ambient cel·lular, està codificada en la seqüència d'ADN igual que ho estan els gens.
Les seqüències reguladores són típicament seqüències curtes presents en les proximitats o en l'interior (freqüentment en introns) dels gens. En l'actualitat, el coneixement sistemàtic d'aquestes seqüències i de com actuen en complexes xarxes de regulació gènica, sensibles a senyals exògens, és molt escàs i està començant a desenvolupar-se mitjançant estudis de genòmica comparada, bioinformàtica i biologia de sistemes. La identificació de seqüències reguladores es basa en part en la recerca de regions no codificants evolutivament conservades.[4] Per exemple, la divergència evolutiva entre el ratolí i l'ésser humà va ocórrer fa 70-90 milions d'anys.[5] Mitjançant estudis de genòmica comparada, alineant seqüències d'ambdós genomes poden identificar-se regions amb alt grau de coincidència, moltes corresponents a gens i altres a seqüències no codificants de proteïnes però de gran importància funcional, atès que han estat sotmeses a pressió selectiva.
Elements ultraconservats
[modifica]Reben aquest nom regions que han mostrat una constància evolutiva gairebé total, major fins i tot que les seqüències codificants de proteïnes, mitjançant estudis de genòmica comparada. Aquestes seqüències generalment s'encavalquen amb introns de gens implicats en la regulació de la transcripció o en el desenvolupament embrionari i amb exons de gens relacionats amb el processament de l'ARN. La seva funció és generalment poc coneguda, però probablement d'extrema importància donat el seu nivell de conservació evolutiva, tal com s'ha exposat en el punt anterior.
En l'actualitat s'han trobat uns 500 segments d'una grandària major a 200 parells de bases totalment conservats (100% de coincidència) entre els genomes d'humà, ratolí i rata, i gairebé totalment conservats en gos (99%) i pollastre (95%).[6]
Pseudogens
[modifica]En el genoma humà s'han trobat així mateix uns 19.000 pseudogens, que són versions completes o parcials de gens que han acumulat diverses mutacions i que generalment no es transcriuen. Es classifiquen en pseudogens no processats (30%) i pseudogens processats (70%).[7]
- Els pseudogens no processats són còpies de gens generalment originades per duplicació, que no es transcriuen per mancar d'una seqüència promotora i haver acumulat múltiples mutacions, algunes de les quals sense sentit (el que origina codons d'aturada prematurs). Es caracteritzen per posseir tant exons com introns.
- Els pseudogens processats, per contra, són còpies d'ARN missatger retrotranscrites i inserides en el genoma. En conseqüència manquen d'introns i de seqüència promotora.
ADN intergènic
[modifica]Com s'ha dit, les regions intergèniques o extragèniques comprenen la major part de la seqüència del genoma humà, i la seva funció és generalment desconeguda. Bona part d'aquestes regions està composta per elements repetitius, classificables com repeticions en tàndem o repeticions disperses, encara que la resta de la seqüència no respon a un patró definit i classificable. Gran part de l'ADN intergènic pot ser un artefacte evolutiu sense una funció determinada en el genoma actual, pel que tradicionalment aquestes regions han estat denominades ADN "escombraria" (Junk DNA), denominació que inclou també les seqüències intròniques i pseudogens. No obstant això, aquesta denominació no és la més encertada donat el paper regulador conegut de moltes d'aquestes seqüències. A més el notable grau de conservació evolutiva d'algunes d'aquestes seqüències sembla indicar que posseeixen altres funcions essencials encara desconegudes o poc conegudes. Per tant, alguns prefereixen denominar-lo "ADN no codificant" (encara que l'anomenat "ADN escombraria" inclou també transposons codificants) o "ADN repetitiu".

ADN repetit en tàndem
[modifica]Són repeticions que s'ordenen de manera consecutiva, de manera que seqüències idèntiques, o gairebé, es disposen unes darrere d'unes altres.
Satèl·lits
[modifica]El conjunt de repeticions en tàndem de tipus satèl·lit comprèn un total de 250 Mb del genoma humà. Són seqüències d'entre 5 i diversos centenars de nucleòtids que es repeteixen en tàndem milers de vegades generant regions repetides amb grandàries que oscil·len entre 100 kb (100.000 nucleòtids) fins a diverses megabases.
Reben el seu nom de les observacions inicials de centrifugacions en gradient de densitat de l'ADN genòmic fragmentat, que reportaven una banda principal corresponent a la major part del genoma i tres bandes satèl·lit de menor densitat. Això es deu al fet que les seqüències satèl·lit tenen una riquesa en nucleòtids Adenosina+Timina superior a la mitjana del genoma i en conseqüència són menys denses.
Hi ha principalment 6 tipus de repeticions d'ADN satèl·lit:[6]
- Satèl·lit 1: seqüència bàsica de 42 nucleòtids. Situat en els centròmers dels cromosomes 3 i 4 i el braç curt dels cromosomes acrocèntrics (en posició distal respecte al clúster codificant d'ARNr).
- Satèl·lit 2: la seqüència bàsica és ATTCCATTCG. Present en les proximitats dels centròmers dels cromosomes 2 i 10, i en la constricció secundària d'1 i 16.
- Satèl·lit 3: la seqüència bàsica és ATTCC. Present en la constricció secundària dels cromosomes 9 i Y, i en posició proximal respecte al clúster d'ADNr del braç curt dels cromosomes acrocèntrics.
- Satèl·lit alfa: seqüència bàsica de 171 nucleòtids. Forma part de l'ADN dels centròmers cromosòmics.
- Satèl·lit beta: seqüència bàsica de 68 nucleòtids. Apareix entorn del centròmer en els cromosomes acrocèntrics i en la constricció secundària del cromosoma 1.
- Satèl·lit gamma: seqüència bàsica de 220 nucleòtids. Pròxim al centròmer dels cromosomes 8 i X.
Minisatèl·lits
[modifica]Estan composts per una unitat bàsica de seqüència de 6-25[6] nucleòtids que es repeteix en tàndem generant seqüències d'entre 100 i 20.000 parells de bases. S'estima que el genoma humà conté uns 30.000 minisatèl·lits.
Diversos estudis han relacionat els minisatèl·lits amb processos de regulació de l'expressió gènica, com el control del nivell de transcripció, l'empalmament (splicing) alternatiu o la impressió genètica (imprinting). Així mateix, s'han associat amb punts de fragilitat cromosòmica atès que se situen pròxims a llocs preferents de trencament cromosòmic, translocació cromosòmica i recombinació meiòtica. Finalment, alguns minisatèl·lits humans (10%) són hipermutables, presentant una taxa mitjana de mutació entre el 0,5% i el 20% en les cèl·lules de la línia germinal, sent així les regions més inestables del genoma humà conegudes fins a la data.
En el genoma humà, aproximadament el 90% dels minisatèl·lits se situen en els telòmers dels cromosomes. La seqüència bàsica de sis nucleòtids TTAGGG es repeteix milers de vegades en tàndem, generant regions de 5-20 kb que conformen els telòmers.
Alguns minisatèl·lits per la seva gran inestabilitat presenten una notable variabilitat entre individus distints. Es consideren polimorfismes multial·lèlics, atès que poden presentar-se en un nombre de repeticions molt variable, i es denominen VNTR (acrònim de Variable number tandem repeat). Són marcadors molt utilitzats en genètica forense, ja que permeten establir una petjada genètica característica de cada individu, i són identificables mitjançant transferència d'RNA i hibridació d'àcids nucleics.
Microsatèl·lits
[modifica]Estan composts per seqüències bàsiques de 2-4 nucleòtids, la repetició dels quals en tàndem origina freqüentment seqüències de menys de 150 nucleòtids. Alguns exemples importants són el dinucleòtid CA i el trinucleòtid CAG.
Els microsatèl·lits són també polimorfismes multial·lèlics, denominats STR (acrònim de Short Tandem Repeats) i poden identificar-se mitjançant PCR, de manera ràpida i senzilla.
S'estima que el genoma humà conté uns 200.000 microsatèl·lits, que es distribueixen més o menys homogèniament, al contrari que els minisatèl·lits, el que els fa més informatius com marcadors.
ADN repetit dispers
[modifica]Són seqüències d'ADN que es repeteixen de manera dispersa per tot el genoma, constituint el 45% del genoma humà. Els elements quantitativament més importants són els LINEs i SINEs, que es distingeixen per la grandària de la unitat repetida.
Aquestes seqüències tenen la potencialitat d'autopropagar-se al transcriure's a un ARNm intermediari, retrotranscriure's i inserir-se en un altre punt del genoma. Aquest fenomen es produeix amb una baixa freqüència, estimant-se que 1 de cada 100-200 nounats porten una inserció nova d'un Alu o un L1, que poden resultar patogènics per mutagènesi insercional, per desregulació de l'expressió de gens pròxims (pels mateixos promotors dels SINE i LINE) o per recombinació il·legítima entre dues còpies idèntiques de diferent localització cromosòmica (recombinació intracromosòmica o inter), especialment entre elements Alu.
| Tipus de repetició | Homo sapiens |
Drosophila melanogaster |
Caenorhabditis elegans |
Arabidopsis thaliana |
|---|---|---|---|---|
| LINE,SINE | 33,4% | 0,7% | 0,4% | 0,5% |
| LTR/HERV | 8,1% | 1,5% | 0% | 4,8% |
| Transposons ADN | 2,8% | 0,7% | 5,3% | 5,1% |
| Total | 44,4% | 3,1% | 6,5% | 10,4% |
SINE
[modifica]Acrònim de l'anglès Short Interspersed Nuclear Elements (Elements nuclears dispersos curts). Són seqüències curtes, generalment d'uns pocs centenars de bases, que apareixen repetides milers de vegades en el genoma humà. Suposen el 13% del genoma humà,[6] un 10% degut exclusivament a la família d'elements Alu (característica de primats).
Els elements Alu són seqüències de 250-280 nucleòtids presents en 1.500.000[6] còpies disperses per tot el genoma. Estructuralment són dímers quasi idèntics, excepte que la segona unitat conté un afegit de 32 nucleòtids, sent major que la primera. Quant a la seva seqüència, tenen una considerable riquesa en G+C (56%),[6] pel que predominen en les bandes R, i ambdós monòmers presenten una cua poliA (seqüència d'adenines) vestigi del seu origen d'ARNm. A més, posseeixen un promotor de l'ARN polimerasa III per a transcriure's. Es consideren retrotransposons no autònoms, ja que depenen per a propagar-se de la retrotranscripció del seu ARNm per una retrotranscriptasa present en el medi.
LINE
[modifica]
Acrònim de l'anglès Long Interspersed Nuclear Elements (Elements nuclears dispersos llargs). Constituïxen el 20% del genoma humà. La família de major importància quantitativa és LINE-1 o L1 que és una seqüència de 6 kb repetida unes 800.000 vegades de manera dispersa per tot el genoma, encara que la gran majoria de les còpies és incompleta al presentar l'extrem 5' truncat per una retrotranscripció incompleta. Així, s'estima que hi ha unes 5.000 còpies completes de L1, només 90 de les quals són actives,[6] estant la resta inhibides per metilació del seu promotor.
La seva riquesa en G+C és del 42%,[6] pròxima a la mitjana del genoma (41%) i es localitzen preferentment a les bandes G dels cromosomes. Posseeixen, a més, un promotor de l'ARN polimerasa II.
Els elements LINE complets són codificants. En concret LINE-1 codifica dues proteïnes:
- Proteïna d'unió a ARN (RNA-binding protein): codificada pel marc de lectura obert 1 (ORF1, acrònim de l'anglès ‘'Open reading Frame 1)
- Enzim amb activitat retrotranscriptasa i endonucleasa: codificada per l'ORF2.
Per tant, es consideren retrotransopsons autònoms, ja que codifiquen les proteïnes que necessiten per a propagar-se. L'ARN polimerasa II present en el medi transcriu el LINE, i aquest ARNm es tradueix en ambdós marcs de lectura produint una retrotranscriptasa que actua sobre l'ARNm generant una còpia d'ADN del LINE, potencialment capaç d'inserir-se en el genoma. Així mateix aquestes proteïnes poden ser utilitzades per pseudogèns processats o elements SINE per a la seva propagació.
Diversos estudis han mostrat que les seqüències LINE poden tenir importància en la regulació de l'expressió gènica, havent-se comprovat que els gens pròxims a LINE presenten un nivell d'expressió inferior. Això és especialment rellevant perquè aproximadament el 80% dels gens del genoma humà conté algun element L1 en els seus introns.[6]
HERV
[modifica]Acrònim de Human Endogenous RetroVirus (retrovirus endogen humà). Els retrovirus són virus el genoma dels quals està compost per ARN, capaços de retrotranscriure's i integrar el seu genoma en el de la cèl·lula infectada. Així, els HERV són còpies parcials del genoma de retrovirus integrats en el genoma humà al llarg de l'evolució dels vertebrats, vestigis d'antigues infeccions retrovirals que van afectar cèl·lules de la línia germinal. Algunes estimacions estableixen que hi ha unes 98.000[8] seqüències HERV, mentre que unes altres afirmen que són més de 400.000. En qualsevol cas, s'accepta que entorn del 5-8% del genoma humà està constituït per genomes antigament virals. La grandària d'un genoma retroviral complet és d'entorn de 6-11 kb, però la majoria dels HERV són còpies incompletes.
Al llarg de l'evolució aquestes seqüències sense interès per al genoma hoste han anat acumulant mutacions sense sentit i delecions que els han inactivat. Encara que la majoria de les HERV tenen milions d'anys d'antiguitat, almenys una família de retrovirus es va integrar durant la divergència evolutiva d'humans i ximpanzés, la família HERV-K(HML2), que suposa entorn de l'1% dels HERV.
Transposons d'ADN
[modifica]Sota la denominació de transposons de vegades s'inclouen els retrotransposons, tals com els pseudogens processats, els SINEs i els LINEs. En tal cas es parla de transposons de classe I per a fer referència als retrotransposons, i de classe II per a referir-se a transposons d'ADN, als quals es dedica el present apartat.
Els transposons d'ADN complets posseïxen la potencialitat d'autopropagar-se sense un intermediari d'ARNm seguit de retrotranscripció. Un transposó conté en gen d'un enzim transposasa, flanquejat per repeticions invertides. El seu mecanisme de transposició es basa a tallar i enganxar, movent la seva seqüència a altra localització distinta del genoma. Els diferents tipus de transposases actuen de manera diferent, havent algunes capaces d'unir-se a qualsevol part del genoma mentre que unes altres s'uneixen a seqüències diana específiques. La transposasa codificada pel mateix transposó ho extreu realitzant dues corts flanquejants en el bri d'ADN, generant extrems cohesius, i ho insereix en la seqüència diana en altre punt del genoma. Una ADN polimerasa emplena els buits generats pels extrems cohesius i una ADN lligasa restablece els enllaços fosfodièster, recuperant la continuïtat de la seqüència d'ADN. Això comporta una duplicació de la seqüència diana entorn del transposó, en la seva nova localització.
S'estima que el genoma humà conté unes 300.000 còpies[6] d'elements repetits dispersos originats per transposons d'ADN, constituint un 3% del genoma. Hi ha múltiples famílies, de les quals cap destacar per la seva importància patogènica per la generació de reordenacions cromosòmiques els elements mariner, així com les famílies MER1 i MER2.
Variabilitat
[modifica]Si bé dos éssers humans del mateix sexe comparteixen un percentatge elevadíssim (entorn del 99,9%[6]) de la seva seqüència d'ADN, el que ens permet treballar amb una única seqüència de referència, petites variacions genòmiques fonamenten bona part de la variabilitat fenotípica interindividual. Una variació en el genoma, per substitució, deleció o inserció, es denomina polimorfisme o al·lel genètic. No tot polimorfisme genètic provoca una alteració en la seqüència d'una proteïna o del seu nivell d'expressió, és a dir, molts són silenciosos i manquen d'expressió fenotípica.
SNPs
[modifica]La principal font de variabilitat en els genomes de dos éssers humans procedeix de les variacions en un sol nucleòtid, coneguts cin SNPs (Sengonal Nucleotide Polimorphisms), en les quals s'han centrat la major part dels estudis. Donada la seva importància, en l'actualitat existeix un projecte internacional (International HapMap Project) per a catalogar a gran escala els SNPs del genoma humà. En aquest context, la denominació de SNP freqüentment es restringeix a aquells polimorfismes d'un sol nucleòtid en els quals l'al·lel menys freqüent apareix en almenys l'1% de la població.
Els SNP són marcadors tetral·lèlics, atès que en teoria en una posició pot haver quatre nucleòtids distints, cadascun dels quals identificaria un al·lel; no obstant això, en la pràctica solen presentar només dos al·lels en la població. S'estima que la freqüència de SNPs en el genoma humà és d'un SNP cada 500-100 parells de bases, dels quals una part rellevant són polimorfismes codificants, que causen la substitució d'un aminoàcid per un altre en una proteïna.
Gràcies a la seva abundància i que presenten una distribució aproximadament uniforme en el genoma, han tingut gran utilitat com marcadors per als mapes de lligament, eina fonamental del Projecte Genoma Humà. A més són fàcilment detectables a gran escala mitjançant l'ocupació de microxips de DNA (comunament coneguts com a microarrays).
Variació estructural
[modifica]Recentment, s'ha començat a estudiar una nova forma de variació en el genoma humà: l'estructural. Aquest tipus de variacions es refereix a duplicacions, inversions, insercions o variants en el nombre de còpies de segments grans del genoma (en general de 1000 nucleòtids o més). Aquestes variants impliquen a una gran proporció del genoma, pel que es pensa que són, almenys, tan importants com els SNPs.[9]
A pesar que aquest camp d'estudi és relativament nou (els primers estudis a gran escala es van publicar en els anys 2004 i 2005), ha tingut un gran auge, fins al punt que s'ha creat un nou projecte per a estudiar aquest tipus de variants en els mateixos individus en els quals es va basar el Projecte HapMap.
Si bé encara queden dubtes sobre les causes d'aquest tipus de variants, cada vegada existeix més evidència a favor que és un fenomen recurrent que encara contínua modelant i creant noves variants del genoma.
Aquest tipus de variacions han potenciat la idea que el genoma humà no és una entitat estàtica, sinó que es troba en constant canvi i evolució.
Malalties genètiques
[modifica]L'alteració de la seqüència d'ADN que constituïx el genoma humà pot causar l'expressió anormal d'un o més gens, originant un fenotip patològic. Les malalties genètiques poden ser causades per mutació de la seqüència d'ADN, amb afectació de la seqüència codificant (produint proteïnes incorrectes) o de seqüències reguladores (alterant el nivell d'expressió d'un gen), o per alteracions cromosòmiques, numèriques o estructurals. L'alteració del genoma de les cèl·lules germinals d'un individu es transmet freqüentment a la seva descendència. Actualment el nombre de malalties genètiques conegudes és aproximadament de 4.000, sent la més comuna la fibrosi quística.
L'estudi de les malalties genètiques freqüentment s'ha englobat dintre de la genètica de poblacions. Els resultats del Projecte Genoma Humà són de gran importància per a la identificació de noves malalties genètiques i per al desenvolupament de nous i millors sistemes de diagnòstic genètic, així com per a la investigació en nous tractaments, inclosa la teràpia gènica.
Mutacions
[modifica]Les mutacions gèniques poden ser:
- Substitucions (canvis d'un nucleòtid per altre): Les substitucions es denominen transicions si suposen un canvi entre bases del mateix tipus químic, o transversions si són un canvi purina (A,G)→pirimidina (C,T) o pirimidina→purina.
- Delecions o insercions: són respectivament l'eliminació o addició d'una determinada seqüència de nucleòtids, de longitud variable. Les grans delecions poden afectar fins i tot a diversos gens, fins al punt de ser apreciables a nivell cromosòmic amb tècniques de citogenètica. Insercions o deleciones d'uns pocs parells de bases en una seqüència codificant poden provocar desplaçament del marc de lectura (frameshift), de manera que la seqüència de nucleòtids de l'ARNm es llegeix de manera incorrecta.
Les mutacions gèniques poden afectar:
- ADN codificant: Si el canvi en un nucleòtid provoca el canvi d'un aminoàcid de la proteïna la mutació es denomina no sinònima. En cas contrari es denominen sinònimes o silencioses (possible perquè el codi genètic és degenerat). Les mutacions no sinònimes així mateix es classifiquen en mutacions amb canvi de sentit (missense) si provoquen el canvi d'un aminoàcid per un altre, mutacions sense sentit (non-sense) si canvien un codó codificant per un codó d'aturada (TAA, TAG, TGA) o amb guany de sentit si succeïx al revés.
- ADN no codificant: Poden afectar seqüències reguladores, promotores o implicades en l'empalmament (splicing). Aquestes últimes poden causar un erroni processament de l'ARNm, amb conseqüències diverses en l'expressió de la proteïna codificada per aquest gen.
Trastorns d'un sol gen
[modifica]Són malalties genètiques causades per mutació en un sol gen, que presenten una herència de tipus mendelià, fàcilment predictible. En la taula es resumeixen els principals patrons d'herència que poden mostrar, les seves característiques i alguns exemples.
| Patró hereditari | Explicació | Exemples |
|---|---|---|
| Autosòmic dominant | Malalties que es manifesten en individus heterozigòtics. És suficient amb una mutació en una de les dues còpies (cada individu posseeix un parell de cada cromosoma) d'un gen perquè es manifesti la malaltia. Els individus malalts generalment tenen un dels seus dos progenitors malalts. La probabilitat de tenir descendència afectada és del 50% atès que cada progenitor aporta un dels cromosomes de cada parell. Freqüentment corresponen a mutacions amb guany de funció (de manera que l'al·lel mutat no és inactiu sinó que posseïx una nova funció que provoca el desenvolupament de la malaltia) o per pèrdua de funció de l'al·lel mutat amb efecte de dosi gènica també conegut com haploinsuficiència. Freqüentment són malalties amb baixa penetrància, és a dir, només una part dels individus que porten la mutació desenvolupen la malaltia. | Malaltia de Huntington, neurofibromatosi 1, síndrome de Marfan, càncer colorectal hereditari no polipòsic |
| Autosòmic recessiu | La malaltia només es manifesta en individus homozigótics recessius, és a dir, aquells en els quals ambdues còpies d'un gen estan mutades. Són mutacions que causen pèrdua de funció, de manera que la causa de la malaltia és l'absència de l'acció d'un gen. La mutació només en una de les dues còpies és compensada per l'existència de l'altra (quan una sola còpia no és suficient s'origina haploinsuficiència, amb herència autosòmica dominant). Habitualment un individu malalt té ambdós progenitors sans però portadors de la mutació (genotip heterozigòtic: Aa). En tal cas un 25% de la descendència estarà afectada. | Fibrosi quística, anèmia falciforme, malaltia de Tay-Sachs, atròfia muscular espinal |
| Dominant lligat al cromosoma X | Les malalties dominants lligades al cromosoma X són causades per mutacions en aquest cromosoma, i presenten un patró hereditari especial. Només unes poques malalties hereditàries presenten aquest patró. Les dones tenen major prevalença de la malaltia que els homes, atès que reben un cromosoma X de la seva mare i altre del seu pare, qualsevol dels quals pot portar la mutació. Els barons en canvi sempre reben el cromosoma Y del seu pare. Així, un baró malalt (xY) tindrà tots els seus fills barons sans (XY) i totes les filles malaltes (Xx), mentre que una dona malalta (Xx) tindrà un 50% de la seva descendència malalta, independentment del sexe. Algunes d'aquestes malalties són letals en barons (xY), de manera que només existeixen dones malaltes (i barons amb síndrome de Klinefelter, XxY). | Hipofosfatèmia, síndrome d'Aicardi |
| Recessiu lligat al cromosoma X | Les malalties recessives lligades al cromosoma X també són causades per mutacions en el cromosoma X. Els barons estan més freqüentment afectats. Un baró portador sempre serà malalt (xY) atès que només posseïx un cromosoma X, que està mutat. La seva descendència seran barons sans (XY) i filles portadores (Xx). Una dona portadora, tindrà una descendència composta per un 50% de filles portadores i un 50% de barons malalts. | Hemofília A, distròfia muscular de Duchenne, daltonisme, distròfia muscular, alopècia androgènica |
| Lligat al cromosoma Y | Són malalties causades per mutació en el cromosoma Y. En conseqüència, només pot manifestar-se en barons, la descendència dels quals serà del 100% de filles sanes i el 100% de fills barons malalts. Donades les funcions del cromosoma Y, freqüentment aquestes malalties només causen infertilitat, que sovint pot ser superada terapèuticament. | Infertilitat masculina hereditària |
| Mitocondrial | Malalties causades per mutació en gens del genoma mitocondrial. Donades les particularitats d'aquest genoma, la seva transmissió és matrilineal (el genoma mitocondrial es transfereix de mares a fills). La gravetat d'una mutació depèn del percentatge de genomes afectats en la població de mitocondris, fenomen denominat heteroplàsmia (en contrast amb heterozigosi), que varia per segregació mitòtica asimètrica. | Neuropatia òptica hereditària de Leber (LHON) |
Trastorns poligènics i multifactorials
[modifica]Altres alteracions genètiques poden ser molt més complexes en la seva associació amb un fenotip patològic. Són les malalties multifactorials o poligèniques, és a dir, aquelles que són causades per la combinació de múltiples al·lel genotípics i de factors exògens, tals com l'ambient o l'estil de vida. En conseqüència no presenten un patró hereditari clar, i la diversitat de factors etiològics i de risc dificulta l'estimació del risc, el diagnòstic i el tractament. Alguns exemples de malalties multifactorials amb etiologia parcialment genètica són:
Alteracions cromosòmiques
[modifica]Les alteracions genètiques poden produir-se també a escala cromosòmica (cromosomopaties), causant severs trastorns que afecten a múltiples gens i que en moltes ocasions són letals provocant avortaments prematurs. Freqüentment són provocades per un error durant la divisió cel·lular, que no obstant això no impedeix la seva conclusió. Les alteracions cromosòmiques reflecteixen una anormalitat en el nombre o en l'estructura dels cromosomes, pel que es classifiquen en numèriques i estructurals. Provoquen fenotips molt diversos, però freqüentment presenten uns trets comuns:
- Retard mental i retard del desenvolupament.
- Alteracions facials i anomalies al capdavant i coll.
- Malformacions congènites, amb afectació preferent d'extremitats, cor, etc.
Numèriques
[modifica]| Aneuploïdia | Freqüència (/1000) |
Síndrome |
|---|---|---|
| Trisomia 21 | 1,5 | de Down |
| Trisomia 18 | 0,12 | d'Edward |
| Trisomía 13 | 0,07 | de Patau |
| Monosomia X | 0,4 | de Turner |
| XXY | 1,5 | de Klinefelter |
| XYY | 1,5 | de l'XYY |
És una alteració del nombre normal de cromosomes d'un individu, que normalment presenta 23 parells de cromosomes (46 en total), sent cada dotació cromosòmica d'un progenitor (diploïdia). Si l'alteració afecta a un només parell de cromosomes es parla d'aneuploïdia, de manera que pot haver un sol cromosoma (monosomia) o més de dos (trisomia, tetrasomia…). Un exemple de gran prevalença és la trisomia 21, responsable de la síndrome de Down. Si per contra l'alteració afecta a tots els cromosomes es parla d'euploïdia, de manera que en teoria l'individu té una sola dotació cromosòmica (haploïdia, 23 cromosomes en total) o més de dues dotacions (triploïdia: 69 cromosomes; tetraploïdia: 92 cromosomes...). En la pràctica les euploïdies causen letalitat embrionària (avortaments) sent molt pocs els nascuts vius, i moren molt primerencament. Les aneuploïdies són majoritàriament letals, excepte les trisomies dels cromosomes 13, 18, 21, X i Y (XXY, XYY), i la monosomia del cromosoma X. En la taula es mostren les freqüències de nascuts vius amb aquestes alteracions.
Estructurals
[modifica]Es denominen així les alteracions en l'estructura dels cromosomes, tals com les grans delecions o insercions, reordenacions del material genètic entre cromosomes... detectables mitjançant tècniques de citogenètica.
- Delecions: eliminació d'una porció del genoma. Alguns trastorns coneguts són la síndrome de Wolf-Hirschhorn per deleció parcial del braç curt del cromosoma 4 (4p), i la síndrome de Jacobsen o deleció 11q terminal.
- Duplicacions: una regió considerable d'un cromosoma es duplica. Un exemple és la malaltia de Charcot-Marie-Tooth tipus 1A, que pot ser causada per duplicació del gen codificant de la proteïna mielínica perifèrica 22 (PMP22) en el cromosoma 17.
- Translocacions: quan una porció d'un cromosoma es transfereix a un altre cromosoma. Hi ha dos tipus principals de translocacions: la translocació recíproca, en la qual s'intercanvien segments de dos cromosomes distints, i la translocació robertsoniana, en la qual dos cromosomes acrocèntrics (13, 14, 15, 21, 22) es fusionen pels seus centròmers (fusió cèntrica).
- Inversions: una part del genoma es trenca i es reorienta en direcció oposada abans de reassociar-se, amb el que aquesta seqüència apareix invertida. Poden ser paracèntriques (si afecten només un braç) o pericèntriques (si la seqüència invertida inclou el centròmer).
- Cromosomes en anells: una porció del genoma es trenca i forma un anell per circularització. Això pot ocórrer amb pèrdua de material o sense pèrdua de material.
- Isocromosomes: cromosomes simètrics, amb els seus dos braç idèntics per deleció d'un dels braços i duplicació de l'altre. El més habitual és l'isocromosoma X, en el qual es perd el braç curt del cromosoma X, originant fenotips de síndrome de Turner.
Les síndromes d'inestabilitat cromosòmica són un grup de trastorns caracteritzats per una gran inestabilitat dels cromosomes, que sofrixen amb gran freqüència alteracions estructurals. Estan associats amb un augment de la malignitat de neoplàsies.
Evolució
[modifica]Els estudis de genòmica comparada es basen en comparança de seqüències genòmiques a gran escala, generalment mitjançant eines bioinformàtiques. Aquests estudis permeten aprofundir en el coneixement d'aspectes evolutius d'escala temporal i espacial molt diversa, des de l'estudi de l'evolució dels primers éssers vius fa milers de milions d'anys o les radiacions filogenètiques en mamífers, fins a l'estudi de les migracions d'éssers humans en els últims 100.000 anys, que expliquen l'actual distribució de les diferents races humanes.
Genòmica comparada (entre espècies)
[modifica]Els estudis de genòmica comparada amb genomes de mamífers suggereixen que aproximadament el 5% del genoma humà s'ha conservat evolutivament en els últims 200 milions d'anys; la qual cosa inclou la gran majoria dels gens i seqüències reguladores. No obstant això, els gens i les seqüències reguladores actualment conegudes suposen només el 2% del genoma, el que suggereix que la major part de la seqüència genòmica amb gran importància funcional és desconeguda. Un percentatge important dels gens humans presenta un alt grau de conservació evolutiva. La similitud entre el genoma humà i el del ximpanzé (Pan troglodytes) és del 98,77%. En terme mitjà, una proteïna humana es diferencia de la seva ortòloga de ximpanzé en tan sols dos aminoàcids, i gairebé un terç dels gens té la mateixa seqüència. Una diferència important entre els dos genomes és el cromosoma 2 humà, que és el producte d'una fusió entre els cromosomes 12 i 13 del ximpanzé.[10]
Una altra conclusió de la comparança del genoma de diferents primats és la notable pèrdua de gens de receptors olfactius que s'ha produït paral·lelament al desenvolupament de la visió en color (tricròmica) durant l'evolució de primats.[11]
Genòmica comparada (entre genomes humans)
[modifica]
Durant dècades les úniques evidències que permetien aprofundir en el coneixement de l'origen i l'expansió de l'Homo sapiens han estat les escasses troballes arqueològics. No obstant això, en l'actualitat, els estudis de genòmica comparada a partir de genomes d'individus actuals de tot el món, estan aportant informació molt rellevant. El seu fonament bàsic consisteix a identificar un polimorfisme, una mutació, que s'assumeix que es va originar en un individu d'una població ancestral, i que ha heretat tota la seva descendència fins a l'actualitat. A més, atès que les mutacions semblen produir-se a un ritme constant, pot estimar-se l'antiguitat d'una determinada mutació sobre la base de la grandària de l'haplotip en el qual se situa, és a dir, la grandària de la seqüència conservada que flanqueja la mutació. Aquesta metodologia es veu complicada pel fenomen de recombinació entre els parells de cromosomes d'un individu, procedents dels seus dos progenitors. No obstant això, hi ha dues regions en les quals no existeix dita inconvenient perquè presenten una herència uniparental: el genoma mitocondrial (d'herència matrilineal), i el cromosoma Y (d'herència patrilineal).
En les últimes dècades, els estudis de genòmica comparada basada en el genoma mitocondrial, i en menor mida en el cromosoma Y, han reportat conclusions de gran interès. En diversos estudis s'ha traçat la filogènia d'aquestes seqüències, estimant-se que tots els éssers humans actuals comparteixen un avantpassat femení comú que va viure a Àfrica fa uns 150.000 anys. Per la seva banda, per raons encara poc conegudes, la major convergència de l'ADN del cromosoma Y estableix que l'avantpassat masculí comú més recent data de fa uns 60.000 anys. Aquests individus han estat batejats com Eva mitocondrial i cromosoma Y d'Adam.
La major diversitat de marcadors genètics i en conseqüència, els haplotips de menor longitud, s'han trobat a Àfrica. Tot la resta de la població mundial presenta només una petita part d'aquests marcadors, de manera que la composició genòmica de la resta de la població humana actual és només un subconjunt de la qual pot apreciar-se a Àfrica. Això induïx a afirmar que un petit grup d'éssers humans (potser entorn d'un miler) va emigrar del continent africà cap a les costes d'Àsia occidental, fa uns 50.000-70.000 anys, segons estudis basats en el genoma mitocondrial. Fa uns 50.000 anys van arribar Austràlia i fa entorn de 40.000-30.000 anys altres subpoblacions colonitzaren Europa occidental i el centre d'Àsia. Així mateix, s'estima que fa 20.000-15.000 anys van arribar el continent americà a través de l'estret de Bering (el nivell del mar era menor durant l'última glaciació, o glaciació de Würm o Wisconsin), poblant Sud-amèrica fa uns 15.000-12.000 anys. No obstant això, aquestes dades només són estimacions, i la metodologia presenta certes limitacions. En l'actualitat, la tendència és combinar els estudis de genòmica comparada basats en l'ADN mitocondrial amb anàlisi de la seqüència del cromosoma Y.
Genoma mitocondrial
[modifica]
És el genoma propi dels mitocondris de cèl·lules eucariotes. El mitocondri és un orgànul subcel·lular essencial en el metabolisme aerobi o oxidatiu de les cèl·lules eucariotes. El seu origen és endosimbiont, és a dir, antigament van ser organismes procariotes independents captats per una cèl·lula eucariota ancestral, amb la qual van desenvolupar una relació simbiòtica. Les característiques del seu genoma, per tant, són molt semblants a les d'un organisme procariota actual, i el seu codi genètic és lleugerament diferent del considerat universal. Per a adaptar-se al nínxol intracel·lular i augmentar la seva taxa de replicació, el genoma mitocondrial s'ha anat reduint substancialment al llarg del seu coevolució, presentant en l'actualitat una grandària de 16.569 parells de bases. Així, la gran majoria de les proteïnes localitzades als mitocondris (1500 en mamífers) estan codificades pel genoma nuclear (al que fan referència tots els apartats anteriors), de manera que molts d'aquests gens van ser transferits del mitocondri al nucli cel·lular durant la coevolució de la cèl·lula eucariota. En la majoria de mamífers, només la femella transmet al zigot els seus mitocondris, pel que presenten, com ja s'ha dit, un patró hereditari matrilineal. En general una cèl·lula humana mitjana conté 100-10.000 còpies del genoma mitocondrial per cada cèl·lula, a raó d'unes 2-10 molècules d'ADN per mitocondri.
El genoma mitocondrial posseeix 37 gens:
- 13 gens codificants de proteïnes: codifiquen 13 polipèptids que formen part dels complexos multienzimátics de la fosforilació oxidativa (sistema OXPHOS). Són 7 subunitatss del Complex I (NADH deshidrogenasa), una subunitat del complex III (citocrom b), 3 subunitats del Complex IV (citocrom oxidasa) i 2 subunitats del Complex V (ATP sintetasa).
- 2 gens ARNr, que codifiquen les dues subunitats de l'ARN ribosòmic de la matriu mitocondrial.
- 22 gens ARNt, que codifiquen els 22 ARN transferents necessaris per a la síntesi proteica en la matriu mitocondrial.
AL contrari del que succeïa amb el genoma nuclear, on només l'1,5% era codificant, en el genoma mitocondrial el 97% correspon a seqüències codificants. És una única molècula d'ADN de doble cadena circular. Una de les hemicadenes rep el nom de cadena pesant o cadena H, i conté 28 dels 37 gens (2 ARNr, 14 ARNt i 12 polipèptids). La hemicadena complementària (cadena lleugera o L) codifica els 9 gens restants. En ambdues cadenes, els gens dels ARNt apareixen distribuïts entre dos gens ARNr o codificants de proteïnes, la qual cosa és de gran importància per al processament de l'ARN mitocondrial.
Vegeu també
[modifica]Referències
[modifica]- ↑ International Human Genome Sequencing Consortium «Finishing the euchromatic sequence of the human genome.». Nature, 431, 7011, 2004, pàg. 931-45. PMID: 15496913.
- ↑ International Human Genome Sequencing Consortium «Initial sequencing and analysis of the human genome.». Nature, 409, 6822, 2001, pàg. 860-921. PMID: 11237011.
- ↑ Watson, JD, Baker TA, Bell SP, Gann A, Levine M, Losick R. (2004). "Ch9-10", Molecular Biology of the Gene, 5th ed., Peason Benjamin Cummings; CSHL Press.
- ↑ Loots G, Locksley R, Blankespoor C, Wang Z, Miller W, Rubin E, Frazer K «Identification of a coordinate regulator of interleukins 4, 13, and 5 by cross-species sequence comparisons.». Science, 288, 5463, 2000, pàg. 136-40. PMID: 10753117. Summary Arxivat 2009-11-06 a Wayback Machine.
- ↑ Nei M, Xu P, Glazko G «Estimation of divergence times from multiprotein sequences for a few mammalian species and several distantly related organisms.». Proc Natl Acad Sci U S A, 98, 5, 2001, pàg. 2497-502. PMID: 11226267.
- ↑ 6,00 6,01 6,02 6,03 6,04 6,05 6,06 6,07 6,08 6,09 6,10 6,11 6,12 Novo Villaverde, F.J.. Genética Humana. Madrid: Pearson, 2007. ISBN 978-84-8322-359-8.
- ↑ Torrents D., Suyama M., Zdobnov E. and Bork p. «A Genome-Wide Survey of Human Pseudogenes.». Genome Research, 13, 12, 2003, pàg. 2559-2567. PMID: 14656963. «Enllaç».
- ↑ Robert Belshaw, (2004). "Long-term reinfection of the human genome by endogenous retroviruses"[Enllaç no actiu] Proc Natl Acad Sci U S A. 2004 April 6; 101(14): 4894–4899
- ↑ Feuk L., Carson A. R. y Scherer S. W. «Structural Variation in the human Genome.». Nature Reviews Genetics, 7, 2, 2006, pàg. 85-97. PMID: 16418744.«Enllaç».
- ↑ "Human chromosome 2 resulted from a fusion of two ancestral chromosomes that remained separate in the chimpanzee lineage" The Chimpanzee Sequencing and Analysis Consortium «Initial sequence of the chimpanzee genome and comparison with the human genome.». Nature, 437, 7055, 2005, pàg. 69-87. PMID: 16136131.
"Large-scale sequencing of the chimpanzee genome is now imminent."Olson M, Varki A «Sequencing the chimpanzee genome: insights into human evolution and disease.». Nat Rev Genet, 4, 1, 2003, pàg. 20-8. PMID: 12509750. - ↑ "Our findings suggest that the deterioration of the olfactory repertoire occurred concomitant with the acquisition of full trichromatic color vision in primates." Gilad Y, Wiebe V, Przeworski M, Lancet D, Pääbo S «Loss of olfactory receptor genes coincides with the acquisition of full trichromatic vision in primates.». PLoS Biol, 2, 1, 2004, pàg. E5. PMID: 14737185.
{1} {2} {3} {4} {5} {6} {7} {8} {9} {10} {11} {12} {13} {14} {15} {16} {17} {18} {19} {20} {21} {22} {X} {Y} |
